HL-SAN耐高鹽核酸酶在高效去除宿主DNA等方面的應用
描述
無論是實驗室規模樣品制備、還是生產規模工藝過程,去除核酸都可以改善工藝流程,如蛋白純化、病毒載體制備、mNGS中樣品處理等過程。而核酸酶的選擇,就顯得特別重要。Heat Labile-Salt Active Nuclease (熱不穩定性耐高鹽核酸酶,HL-SAN) 是一種非特異性內切酶,在高鹽濃度下具有最佳活性;且該酶在多種緩沖液中都有活性,很容易通過還原劑處理而失活。這些特性使HL-SAN在需要去除DNA和RNA的應用中,更為適合。核酸污染,特別是宿主基因組DNA污染,在幾乎所有工藝流程及生物制品的生產中,都是需要重點解決的問題。一方面,宿主DNA的存在,影響目標產物的純化及下游質量分析等。另一方面,宿主DNA殘留能引起機體嚴重的免疫反應,是生物制品的關鍵質量參數之一,FDA及NMPA等對Host Cell DNA (HCD)有嚴格的質控要求。宿主基因組DNA中,組蛋白與DNA形成核小體,核小體緊密折疊形成結構復雜的染色質。組蛋白與DNA間存在強烈的離子作用及疏水作用,再加上特殊的結構,導致宿主DNA很難被準確檢測并清除。已有文獻表明,高鹽濃度下組蛋白與DNA解離相對徹底,這有利于宿主DNA的檢測及去除。但市售的絕大多數核酸酶在高鹽濃度下活性很弱,甚至完全失活。而耐高鹽的HL-SAN成了這類應用的理想選擇。

推薦應用
1. 病原微生物診斷應用,如宏基因組測序(mNGS)樣品去除宿主DNA;
2. 蛋白純化,特別是DNA結合蛋白;
3. 其他需要去除宿主DNA的應用等。
優勢
1. 高鹽條件下,宿主DNA去除更徹底;
2. pI為9.6,容易跟絕大多數蛋白分離;
3. 還原劑存在時,酶易失活,減少對后續流程的影響。
使用指導
1.DNA去除方案從細胞抽提物或細胞裂解液中去除DNA污染,HL-SAN的使用量受到多種因素影響,如細胞類型、裂解液組成、NaCl濃度、Mg2+濃度、裂解液pH等。參考方案見Figure 1。比如,對于1ml細胞裂解樣品,調整到0.3-0.75 M NaCl濃度,加入1000 U HL-SAN,15℃-37℃溫度條件下孵育30-60min,或者4℃過夜。Mg2+是必須的。

Figure 1. 不同樣品、不同去除要求下,HL-SAN的推薦濃度及反應條件。(DNA removal的要求是指通過瓊脂糖凝膠電泳看不到條帶。Decontamination的去除要求是對23S rDNA進行qPCR檢測為陰性。)
2.滅活滅活是通過添加還原劑(TCEP或DTT)來實現的,推薦用TCEP(因為TCEP在磷酸鹽溶液中不穩定,特別在中性pH下)。不同工藝流程中,通過改變孵育時間、溫度和還原劑的濃度等來滅活該酶。一般情況下, 25-37℃反應5-10分鐘,可以滅活99%以上的酶。為了避免酶恢復活性、再次激活,保持低濃度還原劑,如0.1-0.5 mM DTT或TCEP,或延長滅活反應時間。
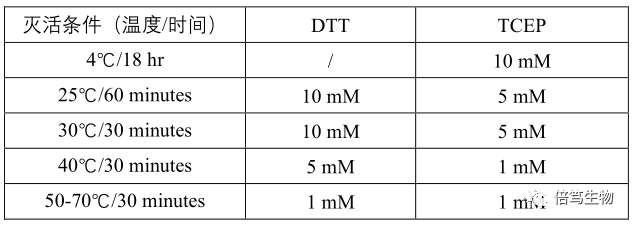
Figure 2. HL-SAN的滅活反應條件
應用實例:
高效去除人體DNA (99.99%)干擾、快速檢測下呼吸道感染(LRIs)病原呼吸道感染(RIs)病原的快速檢測一直是醫療中亟待解決的問題。RIs樣本類型眾多并且病原含量差別很大,同時帶有大量的人體細胞,因此搭建一套快速、高效的樣本處理系統對于通過高通量測序進行病原檢測至關重要。2019年Nature Biotechnology文章報道了如下流程。為了盡可能去除宿主DNA、提高檢測靈敏度,在優化實驗的人體宿主細胞去除、微生物DNA提取和nanopore測序等三個步驟都做了對應的優化,比如宿主DNA去除時找到了合適濃度的saponin (2.2%),HL-SAN步驟由兩步減為一步,清洗步驟縮減為2次。最終,從拿到樣本到建庫完成縮短至6hr,且在最終檢測中達到了96.6%的敏感性和100%的特異性。
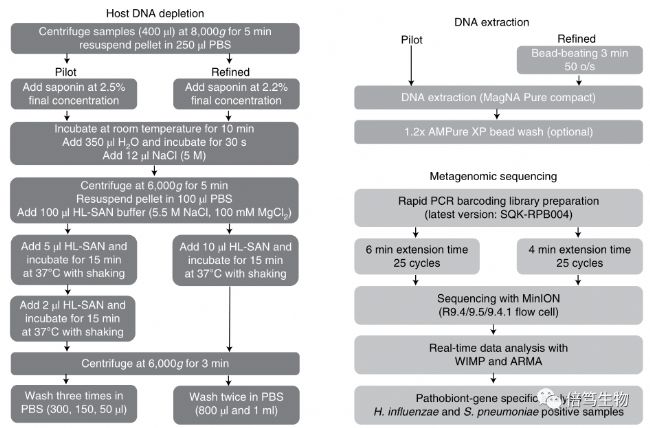
Figure 3. Nanopore宏基因組快檢流程
酶學特征
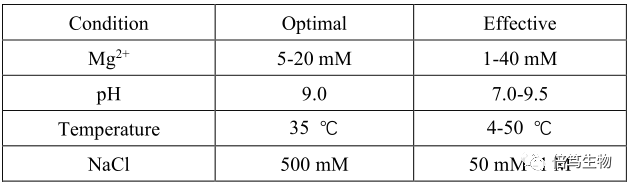
來源:Pichia pastoris酶比活:≥1.75 x 10^5 Units/mg酶活定義:在37℃,25mM Tris-HCl,pH8.5 (25℃),5mM MgCl2,500mM NaCl反應體系中,一個單位的HL-SAN在30分鐘內消化50µg/ml的小牛胸腺DNA(Sigma, D-1501),產生OD260nm處1A的吸光值變化。特異性:非特異性內切核酸酶,能夠將單鏈及雙鏈的DNA及RNA,消化成5個堿基為主的寡核苷酸oligos。酶活條件:(Effective是指活性在最優活性10%以上的條件范圍)
References
1. Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratoryinfection. Charalampous T, Kay GL, O’Grady J. Nature Biotechnology. 2019;37: 783–792.
2. A Structured Workflow for Mapping Human Sin3 Histone Deacetylase ComplexInteractions Using Halo-MudPIT Affinity-Purification Mass Spectrometry. BanksCAS, Thornton JL, Washburn MP. Molecular & Cellular Proteomics. 2018;17 (7): 1432-1447.
3. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibriosalmonicida. Williamson A, Pedersen H. Protein Expr Purif. 2014; 97: 29-36.