motif分析工具——MEME-CHIP
揭秘基因調控的鑰匙:深度解析MEME-CHIP,你的motif分析神器
你是否好奇基因是如何被精確調控的?DNA中的“密碼片段”——motif,正是轉錄因子結合的關鍵位點,它們如同基因開關,控制著生命活動的節奏。今天,我們將帶你認識一款強大的motif分析工具MEME-CHIP,從功能亮點到實操指南,一文掌握!
一、Motif分析:為什么選擇MEME-CHIP?
Motif是DNA、RNA或蛋白質中頻繁出現的短序列模式,能通過結合其他分子調控基因表達。目前主流工具包括HOMER(Homer Software and Data Download)和MEME套件(Introduction - MEME Suite),而MEME-CHIP憑借三大優勢脫穎而出:
你是否好奇基因是如何被精確調控的?DNA中的“密碼片段”——motif,正是轉錄因子結合的關鍵位點,它們如同基因開關,控制著生命活動的節奏。今天,我們將帶你認識一款強大的motif分析工具MEME-CHIP,從功能亮點到實操指南,一文掌握!
一、Motif分析:為什么選擇MEME-CHIP?
Motif是DNA、RNA或蛋白質中頻繁出現的短序列模式,能通過結合其他分子調控基因表達。目前主流工具包括HOMER(Homer Software and Data Download)和MEME套件(Introduction - MEME Suite),而MEME-CHIP憑借三大優勢脫穎而出:
- 精準發現新motif:基于多重期望最大化(EM)算法,無需依賴已知數據庫即可高效識別全新motif。
- 可視化交互體驗:結果頁面支持motif logo圖、反向互補序列查看,并一鍵跳轉至關聯工具(如CentriMo、SpaMo)的深度分析(圖2)。
- 靈活使用場景:提供在線版(圖3)和本地版,支持大規模數據分析,且官網文檔詳盡,小白也能輕松上手。
對比HOMER:
- HOMER依賴已知數據庫,新motif發現能力較弱;
- 結果展示以靜態列表為主,交互性不足;
- 僅支持本地部署,工具生態有限。
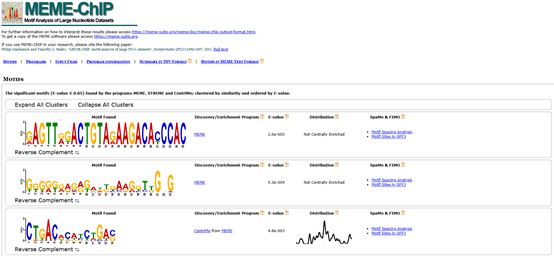

圖 1 MEME-CHIP網頁結果展示

圖 2 MEME官網主頁
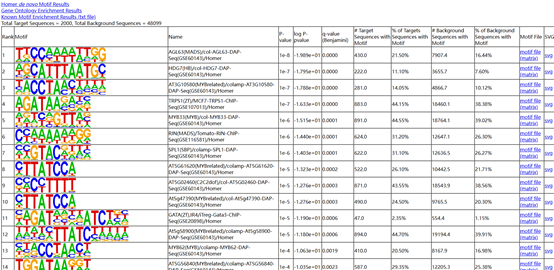
圖 3 HOMER網頁結果頁面展示
二、MEME-CHIP核心功能一覽
作為MEME套件的明星工具,MEME-CHIP在ChIP-seq、ATAC-seq等分析中廣泛應用,六大功能直擊研究痛點:
- 全新motif挖掘:通過MEME和STREME算法,在序列中心區域(默認100bp)鎖定潛在motif。
- 富集區域定位:CentriMo快速篩選顯著富集的motif。
- 數據庫比對:Tomtom將新motif與JASPAR等數據庫匹配,揭示潛在功能。
- 智能分組:自動聚類相似motif,簡化結果解讀。
- 空間規律分析:SpaMo探索motif間的間距分布模式。
- 可視化支持:生成GFF文件,一鍵在基因組瀏覽器中展示結合位點。
三、手把手教你運行MEME-CHIP
本地版使用指南(Linux系統)
1. 常用參數解析
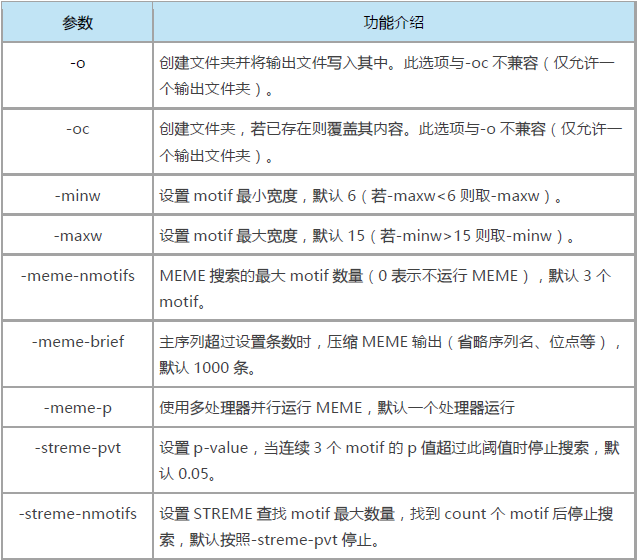
2. 實戰命令示例
meme-chip -oc meme -meme-p 10 -meme-brief 2000 -meme-nmotifs 10 -minw 6 -maxw 20 -streme-nmotifs 10 meme.fasta
這條命令通過指定輸出目錄(-oc meme)、使用10個處理器加速MEME運行(-meme-p 10)、限制序列數量(-meme-brief 2000)、設置MEME搜索的最大motif數量(-meme-nmotifs 10)以及motif的最小(-minw 6)和最大寬度(-maxw 20),并限制STREME查找的motif數量(-streme-nmotifs 10),高效地從輸入序列文件meme.fasta中識別和分析motif。
該命令為主程序,還會有諸多子程序命令運行(圖4)如富集centrimo,spamo分析motif建的間距關系。

圖 4 Meme- CHIP子命令
命令行解析:
- -oc meme:指定輸出目錄為meme
- -meme-p 10:使用10個處理器運行
- -meme-brief 2000:設置序列共2000條
- -meme-nmotifs 10:設置MEME搜索motif最大數量為10個
- -minw 6:設置motif最小寬度為6
- -maxw 20:設置motif最大寬度為20
- -streme-nmotifs 10:設置STREME查找motif最大數量為10個
- meme.fasta:是輸入的序列文件
四、結果解讀:從數據到生物學意義

圖 5 MEME-CHIP輸出結果目錄(所有網頁結果都可點擊對應按鈕“?”查看詳細介紹)
運行完成后,輸出目錄包含以下核心文件:
- meme-chip.html:交互式總覽頁面,整合所有分析結果,支持一鍵跳轉。
- summary.tsv:結構化摘要文件,可直接用Excel打開或腳本解析。
- combined.meme:MEME格式的motif文本文件,包含位點信息。
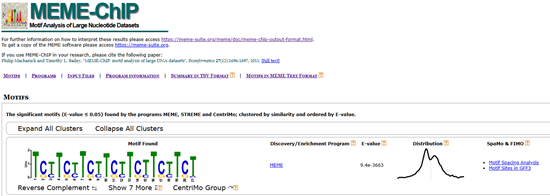
圖 6 meme-chip.html界面

圖 7 導航欄,點擊可跳轉對應部分
導航欄鏈接介紹:
- MOTIFS:查看聚類后的motif及其logo圖、E值、反向互補序列。
- PROGRAMS:運行的各程序的對應命令行。
- INPUT FILES:輸入的文件信息。
- Program information:Linux運行的命令行。
- Summary in TSV Format:tsv格式的結果,點擊“?”可查看各列解釋。
- Motifs in MEME Text Format:text格式結果,點擊“?”可查看文件解釋。
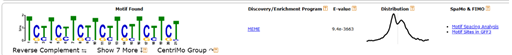
圖 8 motif詳細信息
motif展示信息介紹:
- Discovery/Enrichment Program:對應的是motif發現/富集工具,點擊可跳轉對應工具的分析結果網頁。
- E-value:顯著性E-value。
- Distribution:為Centrimo分析發現序列與motif最佳匹配的分布圖,點擊會跳轉Centrimo分析結果網頁。
- SpaMo & FIMO:展示的是SpaMo和FIMO的分析結果,點擊Motif Spacing Analysis跳轉Spamo分析網頁,點擊Motif Sites in GFF3展示FIMO分析結果中的GFF3文件。
- Reverse Complement:展示motif反向互補序列。
- Show 7 More:展示該類所有motif,點擊“?”,可了解聚類分析過程。
- Centrimo Group:點擊跳轉Centrimo分析結果,及上面提到的centrimo_out中的內容。
主要文件夾網頁內容:
1. centrimo_out為CentriMo的輸出結果目錄,CentriMo是motif富集工具,用于分析在輸入序列上出現富集的已知motif。
1. centrimo_out為CentriMo的輸出結果目錄,CentriMo是motif富集工具,用于分析在輸入序列上出現富集的已知motif。
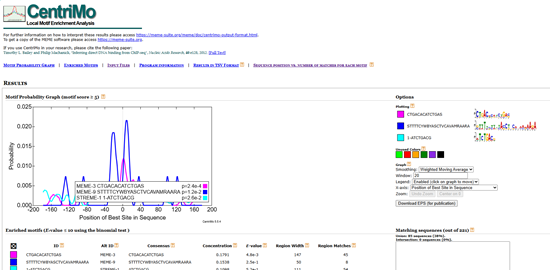
圖 9 centrimo.html界面
- ID:motif的名稱。
- Alt ID:motif的唯一標識符,來源于motif數據庫文件。
- Consensus:基于motif頻率矩陣計算的共識序列。
- E-value:表示在輸入序列中,至少有一個區域與motif的最佳匹配程度相同的預期數量。E值是調整后的 p 值乘以輸入文件中moif的數量。
- Region Width:最富集區域的寬度。
- Region Matches:序列中匹配到該motif的數量。
- 右側Options為交互式界面可調整左側基因概率曲線圖的樣式。
- 右側Matching sequences展示所有選定motif中最顯著區域內至少有一個最佳匹配的序列標識符。“交集”(Intersection)子標題給出了文本框中的標識符數量及其占輸入序列總數的百分比;“并集”(Union)子標題列出了在任何選定motif的最顯著區域內至少有一個最佳匹配的序列數量及其占輸入序列總數的百分比。
2. fimo_out是fimo工具掃描motif后的輸出結果,數字標號對應的是motif編號,如圖10所示,fimo.html中主要為各motif的詳細信息,如寬度,起始位置等。

圖 10 fimo.html界面
3.meme_out是MEME-CHIP重要輸出文件之一,如圖11,主要有各個查找到的motif的logo圖,位置信息等。
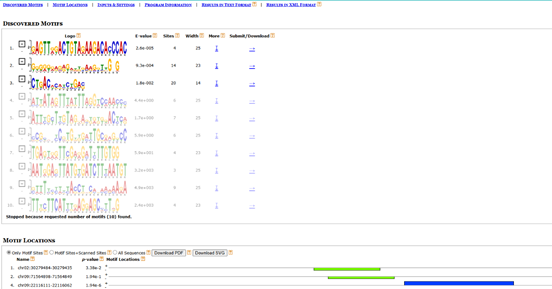
圖 11 meme.html界面
4. spamo_out是SpaMo工具的輸出目錄,用于分析motif之間的間距關系,如圖12中的Spacing Analysis使用logo圖形式展示了間距分析結果。
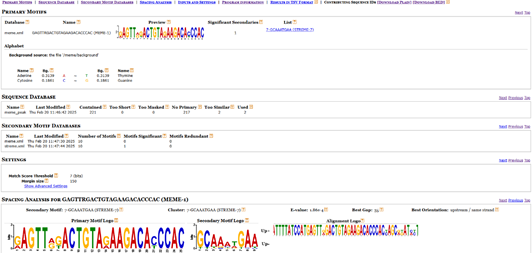
圖 12 spamo.html界面
5.streme_out是STREME工具的輸出目錄,專門用于在輸入序列中發現短的、重復的motif,如圖13所示,所展示logo圖明顯較其他分析工具的motif要短。

圖 13 streme.html界面
五、學術影響力:MEME-CHIP的科研背書
核心文獻:
核心文獻:
- The MEME Suite(截止2025年2月21日星期五,引用3725次):奠定工具生態基礎。
- MEME-ChIP: motif analysis of large DNA datasets(截止2025年2月21日星期五,引用1830次):專為高通量數據優化。
- 適用場景:從轉錄因子結合模式到基因調控網絡構建,MEME-CHIP助你解鎖數據深層價值。
參考文獻
[1]Bailey T L, Johnson J, Grant C E, et al. The MEME suite[J]. Nucleic acids research, 2015, 43(W1): W39-W49.
[2]Machanick P, Bailey T L. MEME-ChIP: motif analysis of large DNA datasets[J]. Bioinformatics, 2011, 27(12): 1696-1697.
[1]Bailey T L, Johnson J, Grant C E, et al. The MEME suite[J]. Nucleic acids research, 2015, 43(W1): W39-W49.
[2]Machanick P, Bailey T L. MEME-ChIP: motif analysis of large DNA datasets[J]. Bioinformatics, 2011, 27(12): 1696-1697.
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com