單細胞轉錄組Smart-seq 2和10x Genomics Chromium技術原理及應用
應用單細胞測序技術研究科學問題越來越普遍,當下應用最火熱的是10x Genomics公司的Chromium解決方案(以下簡稱10X)。但是基于2017年Christoph Ziegenhain et al[1]對6種單細胞轉錄組技術及2019 Broad研究所的團隊對7種單細胞RNA測序方法進行了比較[2],某些特殊或者少量細胞樣本的單細胞轉錄組研究中,Smart-seq2技術還是一項研究利器。
Smart-seq2與10x技術的區別與應用的科研場景有哪些呢?北京大學張澤民團隊在2019年發表的預印文章“Direct Comparative Analysis of 10x Genomics Chromium and Smart-seq2”中給予了答案。
那么今天小編再次對Smart-seq 2與10x Genomics Chromium的技術原理和應用進行闡明。
Smart-Seq簡介
Smart-Seq (Switching mechanism at 5' end of the RNA transcript)于2012年發表[3],2013年發表了其改進技術的應用Smart-Seq2[4],2014年Smart-Seq2 protocol發表[5]。Smart-Seq2 對原始的Smart-Seq實驗流程進行了多項改進優化,它不再需要純化步驟,可大大提高產量,最重要的改進是下面兩項:
Smart-seq2與10x技術的區別與應用的科研場景有哪些呢?北京大學張澤民團隊在2019年發表的預印文章“Direct Comparative Analysis of 10x Genomics Chromium and Smart-seq2”中給予了答案。
那么今天小編再次對Smart-seq 2與10x Genomics Chromium的技術原理和應用進行闡明。
Smart-Seq簡介
Smart-Seq (Switching mechanism at 5' end of the RNA transcript)于2012年發表[3],2013年發表了其改進技術的應用Smart-Seq2[4],2014年Smart-Seq2 protocol發表[5]。Smart-Seq2 對原始的Smart-Seq實驗流程進行了多項改進優化,它不再需要純化步驟,可大大提高產量,最重要的改進是下面兩項:
- TSO 3'端最后一個鳥苷酸替換為鎖核酸LNA(locked nucleic acid)。LNA單體的熱穩定性增強,其退火溫度增強非模板cDNA的3'延伸能力。
- 甜菜堿(一種具有兩個重要作用的甲基供體:它會增加蛋白質的熱穩定性,并通過破壞DNA螺旋來降低甚至消除了DNA熱融變對堿基對組成的依賴性)與較高的MgCl2濃度結合使用。解決某些RNA形成二級結構(例如發夾或環)由于空間位阻,可能導致酶終止鏈延長的問題。
Smart技術是基于高保真的反轉錄酶、模板轉換和前置放大來增加cDNA得率,實驗流程2天,得到的是全長轉錄本。該方法有較好的覆蓋范圍,可檢測到稀有轉錄本,不需要額外的專業設備,因此應用范圍較廣。
Smart-Seq2建庫原理
Smart-Seq2建庫原理
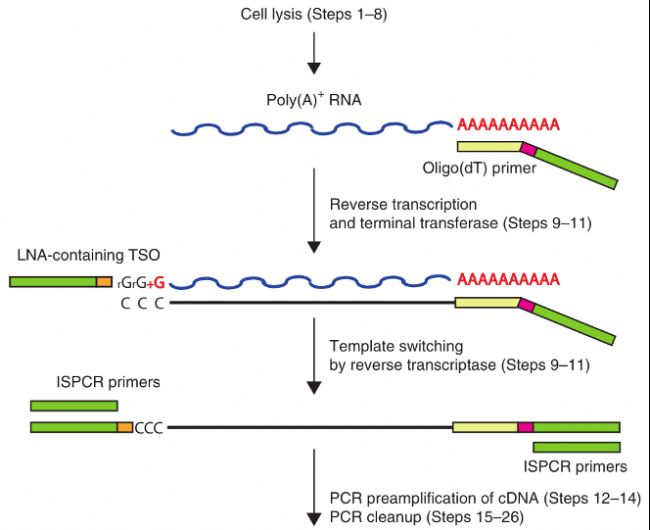
- 單細胞分選:Smart-seq2使用流式細胞儀或顯微操作進行細胞分選,體積不超過0.5 ul。
- 細胞裂解:將分離細胞直接轉移到細胞裂解液中進行細胞裂解。
- 反轉錄( 一鏈合成 ):使用Oligo(dT) primer 對帶有polyA尾的RNA( 主要mRNA )進行反轉錄。由于使用了特殊活性反轉錄酶( Moloney Murine Leukemia Virus )進行反轉錄,所以會在cDNA鏈3'端加上三個C。
- 模板置換( 二鏈合成 ):該步使用TSO ( template-switching oligo )引物合成了cDNA的二鏈,從而置換了與一鏈cDNA互補的RNA。要注意的是TSO 3'端有三個G能與一鏈3'端的三個C互補,而最末端的+G是一個修飾過的G,能增加TSO的熱穩定性,以及其與一鏈cDNA游離的3’端的互補的能力。
- PCR擴增:該步進行輕度的cDNA富集,將cDNA擴增至ng級即可。
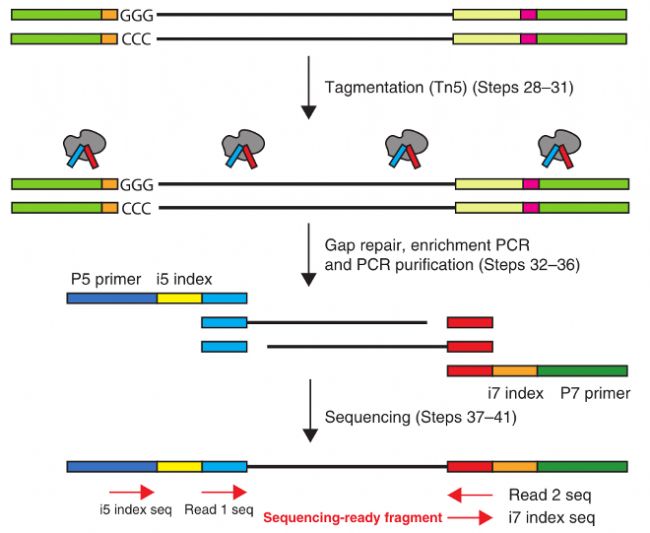
- 標記:利用改造后的高活性Tn5轉座酶對DNA進行打斷的同時將接頭添加到cDNA的兩端。標記完成后的DNA片段通常在200-600bp。
- PCR富集及上機測序:在進行最后一次PCR擴增后,即可上機測序。
10x Genomics單細胞技術
ChromiumTM Single Cell 3' Solution是基于10 X Genomics平臺,能夠一次性分離、并標記5000–10000個單細胞,并能在單細胞水平進行檢測的技術。該方法基于微流控技術(Microfluidics-based approaches),與Smart有相似的分子生物學原理,運用了模板轉換技術,但與Smart的細胞捕獲和通量不同。droplet-based方法是將單個細胞包裹在一個小油滴中(含有barcode和RT primer)反轉錄成cDNA,然后油滴破裂釋放cDNA,統一進行文庫構建,增大了實驗通量,但需要專門的實驗設備。
10x Genomics建庫原理
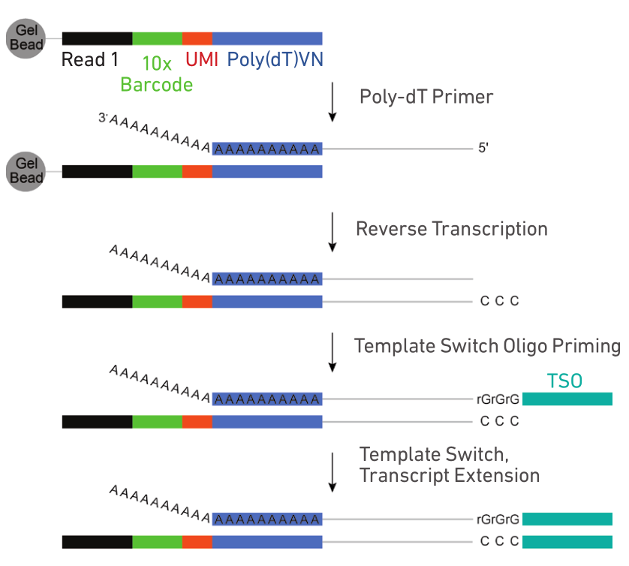
- 制備好的細胞懸液、10x barcode凝膠磁珠和油滴分別加入到Chromium Chip B的不同小室,經由微流體“雙十字”交叉系統形成GEM。為了獲得單細胞反應體系,細胞懸液濃度建議控制在700-1200細胞/ul,因此產生的90-99% GEM不含有細胞,剩下的大部分GEM含有一個細胞。
- 單個GEM依次形成后再全部混合,細胞裂解,凝膠珠自動溶解釋放大量引物序列。
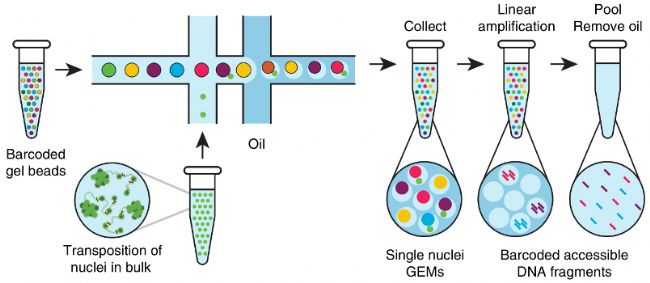
3. 釋放的引物中包含30nt poly dT反轉錄引物,帶有polyA的RNA被反轉錄為帶有10x Barcode和UMI信息的cDNA一鏈,再以SMART方式完成二鏈合成。
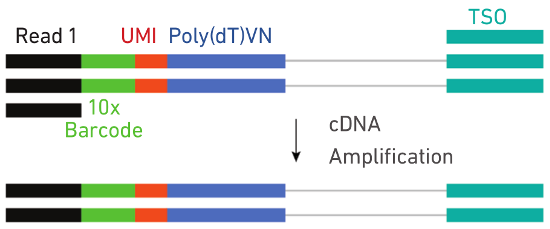
4. 油滴破碎,磁珠純化cDNA一鏈,然后PCR擴增cDNA。
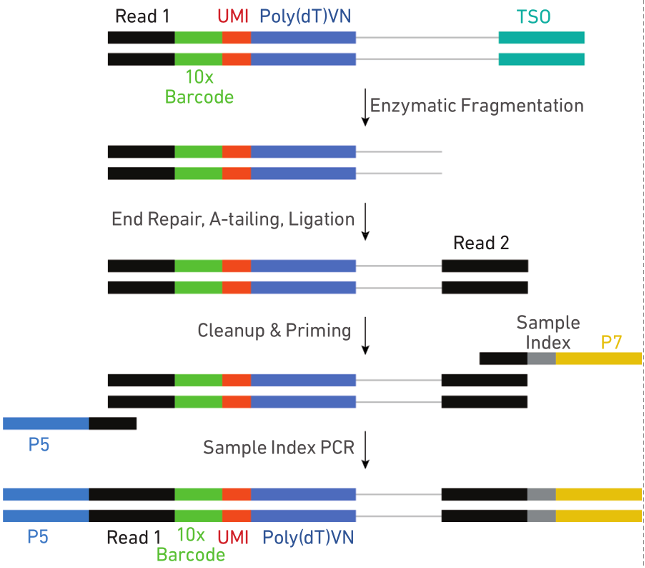
5. cDNA擴增完成后酶切片段化并磁珠篩選最適片段,通過末端修復、加A、接頭連接Read2測序引物,再以PCR方式構建含有P5和P7接頭的cDNA文庫即可。
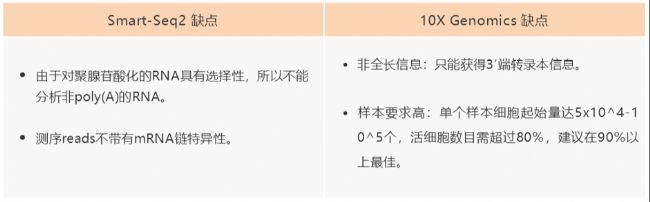
Smart-Seq2 和 10X Genomics 優點與限制

數據差異
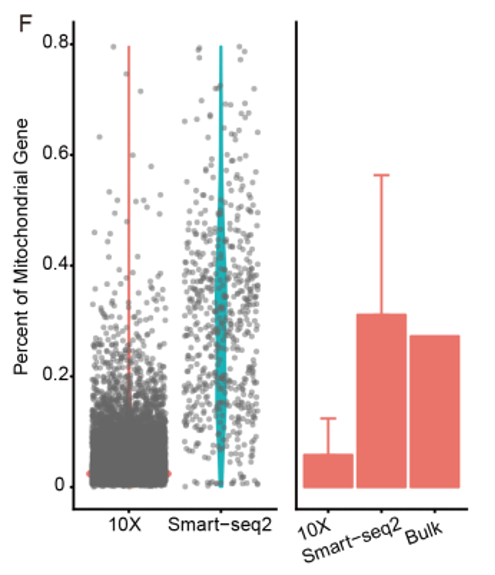
張澤民團隊通過直接比較兩個平臺上來自相同CD45細胞樣本的scRNA-seq數據,廣泛系統地分析評估了各自數據特征。 Smart-seq2在細胞中檢測到更多的基因和更為復雜的數據集,尤其是豐度較低的轉錄本以及可變剪切的轉錄本,但捕獲了更高比例的線粒體基因。 Smart-seq2數據的組合也更類似于bulk RNA-seq數據。
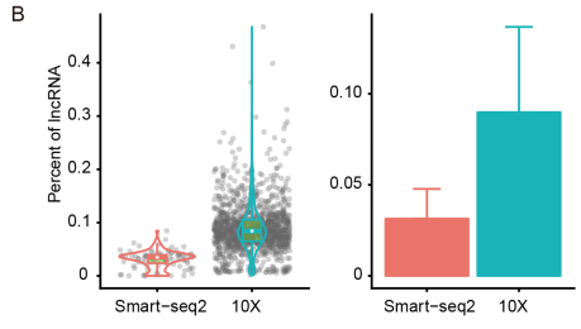
盡管都是基于poly(A)富集策略,兩個平臺檢測到的所有轉錄本中約有10-30%來自非編碼基因,而lncRNA在10x中所占的比例更高。
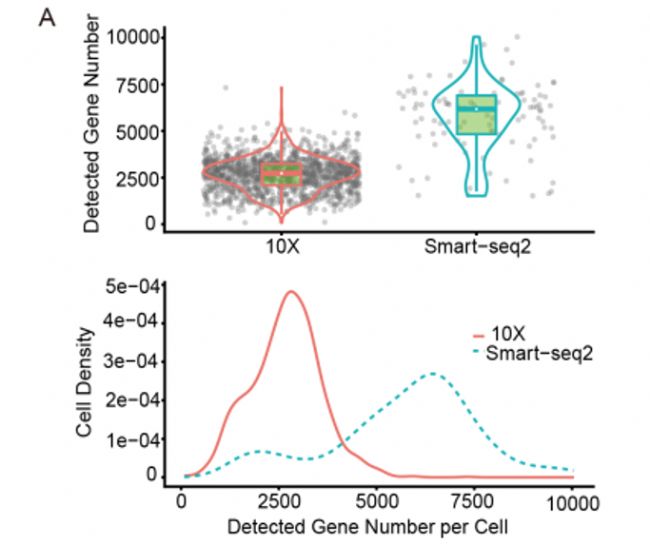
Smart-seq2具有更高的敏感度,檢測到的表達基因數目遠高于10x的檢測到的表達基因數目。
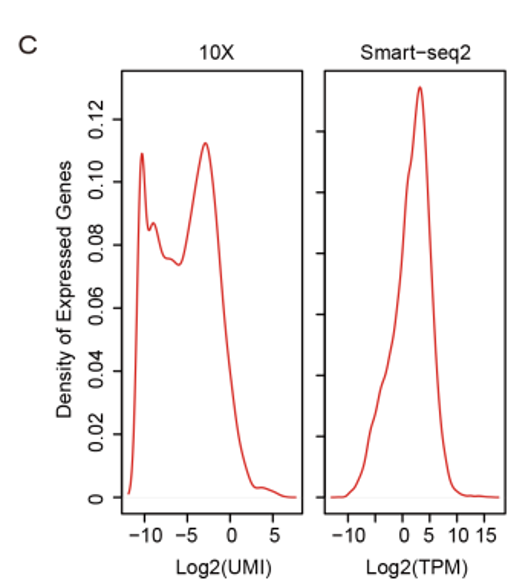
對于檢測到的基因,Smart-seq2數據呈單峰分布,在所有細胞中檢測到的低表達基因很少。相比之下,10x的數據由于有大量的接近零表達的基因,呈現出明顯的雙峰分布,說明10x數據中mRNA在很低的表達水平上有較高的噪聲或隨機捕獲。
基于10x的數據顯示出更嚴重的dropout問題(dropout是scRNA-Seq數據的一大特點,就是很多基因在某些細胞根本就檢測不到表達,但是在另外的細胞卻檢測為高表達。通常認為這個dropout是因為在文庫構建的過程中,有部分基因沒有被成功的反轉錄。),尤其是對于表達水平較低的基因。
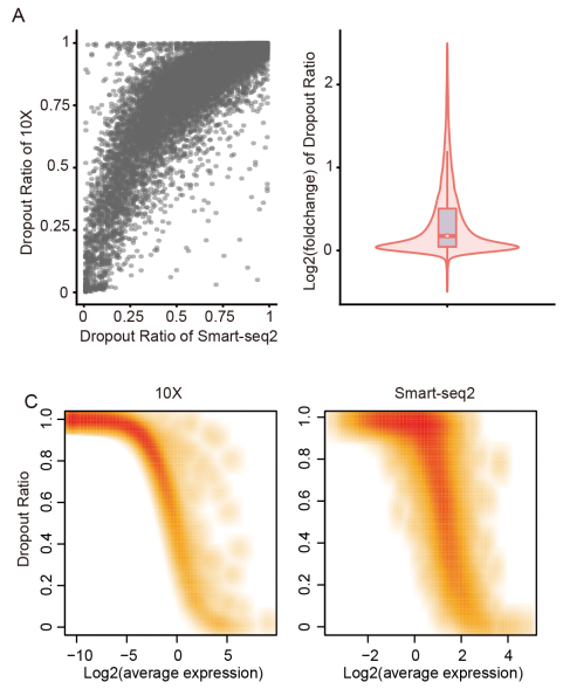
10x 技術測序數據由于技術特色在mRNA的3’端有著較強的bias,而Smart-seq2技術則在gene上有著更為均一的分布。同時, 10x技術數據在splicing分析層面并不適用。

但是,由于10x數據能夠覆蓋大量細胞,因此可以更好地檢測稀有細胞類型。此外,每個平臺檢測到的細胞簇之間差異表達基因的不同集合,表明這些技術是具有互補性的。
Smart-seq2技術案例
Björklund 2016年利用Smart-seq2單細胞轉錄組測序揭示人CD127+先天淋巴細胞的異質性[6]。先天淋巴細胞(ILCs)是一種免疫細胞,它通過釋放可溶性的因素調節對病毒、細菌和寄生蟲的早期免疫反應。這些細胞可以根據細胞表面的轉錄因子及細胞因子被分為三個亞型,每種亞型都有不同的功能。利用流式細胞儀分選患有阻塞性睡眠呼吸暫停綜合癥患者的扁桃體組織中的ILCs(Lin−CD127+)和NK cells(CD45+Lin−CD127−NKG2A+CD56+CD16− )。采用Smart-seq2進行單細胞轉錄組擴增建庫;Illumina HiSeq 2000 3M reads /樣本進行測序分析。采用PCA(主成分分析)和t-SNE對ILC中847個表達的基因進行分型分析,將細胞分為4類:ILC1、ILC2、ILC3、NK cells。應用SCDE軟件包對4類細胞的差異基因進行分析,找出共有及特有差異基因,結果發現,CD127+ILCs中共有的差異基因的表達量比NK細胞中明顯要高。 ILC1、 ILC2、ILC3差異基因表達分析,結果表明,ILC1 cells共有79個上調表達基因,參與干擾素γ的調控,ILC2 cells共有58個上調表達的基因,在前列腺素、Notch信號通路及環境感應中發揮作用,ILC3 cells共有371個上調表達的基因,根據GO注釋有85個與免疫相關,且存在未知功能的基因。
Smart-seq2技術案例
Björklund 2016年利用Smart-seq2單細胞轉錄組測序揭示人CD127+先天淋巴細胞的異質性[6]。先天淋巴細胞(ILCs)是一種免疫細胞,它通過釋放可溶性的因素調節對病毒、細菌和寄生蟲的早期免疫反應。這些細胞可以根據細胞表面的轉錄因子及細胞因子被分為三個亞型,每種亞型都有不同的功能。利用流式細胞儀分選患有阻塞性睡眠呼吸暫停綜合癥患者的扁桃體組織中的ILCs(Lin−CD127+)和NK cells(CD45+Lin−CD127−NKG2A+CD56+CD16− )。采用Smart-seq2進行單細胞轉錄組擴增建庫;Illumina HiSeq 2000 3M reads /樣本進行測序分析。采用PCA(主成分分析)和t-SNE對ILC中847個表達的基因進行分型分析,將細胞分為4類:ILC1、ILC2、ILC3、NK cells。應用SCDE軟件包對4類細胞的差異基因進行分析,找出共有及特有差異基因,結果發現,CD127+ILCs中共有的差異基因的表達量比NK細胞中明顯要高。 ILC1、 ILC2、ILC3差異基因表達分析,結果表明,ILC1 cells共有79個上調表達基因,參與干擾素γ的調控,ILC2 cells共有58個上調表達的基因,在前列腺素、Notch信號通路及環境感應中發揮作用,ILC3 cells共有371個上調表達的基因,根據GO注釋有85個與免疫相關,且存在未知功能的基因。
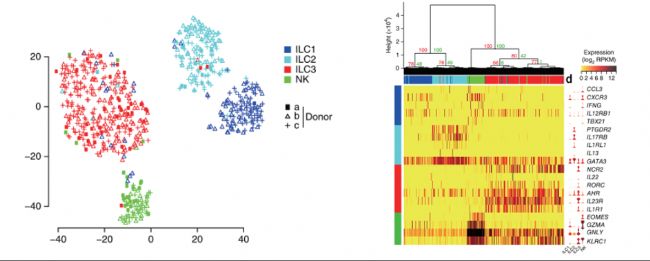
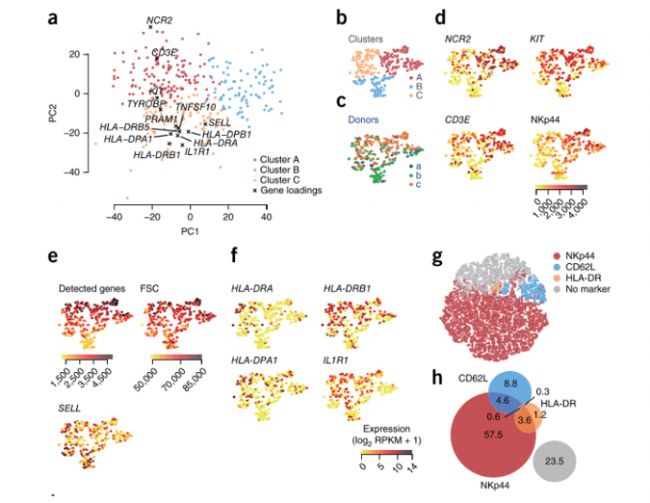
采用PCA和t-SNE分析ILC3中1,958個注釋的免疫基因,將ILC3分為3個亞型:Cluster A、Cluster B、Cluster C, 通過對這3種亞型細胞的分析,發現了新的免疫細胞CD62L+ ILC3。本研究通過對648個單細胞進行單細胞轉錄組的分析,應用t-SNE細胞分型、SCDE基因差異表達分析,在ILC3中發現新的免疫細胞CD62L+ ILC3。
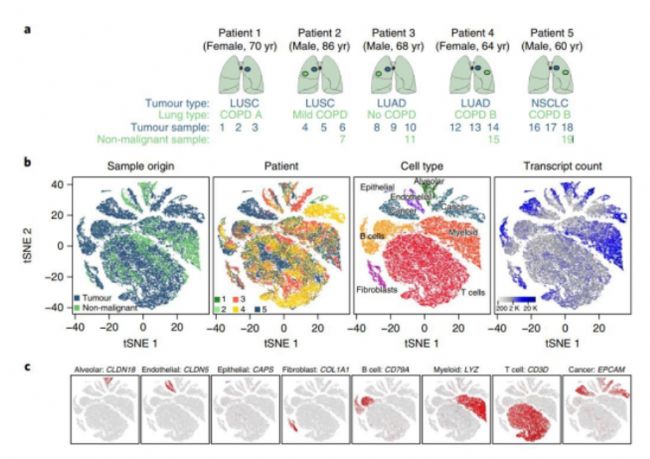
10x Genomics技術案例
比利時的研究團隊通過對數以千計的健康與癌變肺細胞的研究,創建了史上第一個完整的肺癌細胞圖譜。研究結果表明,肺癌較先前認識復雜得多,其包含了52種不同類型的基質細胞,這些新信息可用于開發新的肺癌治療途徑[7]。
Smart-seq2與10x Genomics聯合技術案例
2019年10月31日,北京大學生物醫學前沿創新中心(BIOPIC)、生命科學學院、北京未來基因診斷高精尖創新中心(ICG)張澤民教授、任仙文副教授聯合首都醫科大學附屬北京世紀壇醫院彭吉潤教授以及勃林格殷格翰公司劉康博士,在Cell上發表了題為Landscape and Dynamics of Single Immune Cells in Hepatocellular Carcinoma的研究論文[8]。該研究結合10x Genomics和SMART-seq2兩種單細胞RNA測序技術,對肝癌患者多個組織的免疫細胞做出了系統性的刻畫,分析了免疫細胞動態遷移和狀態轉化的特征,探索了它們在肝癌治療上的潛在價值。
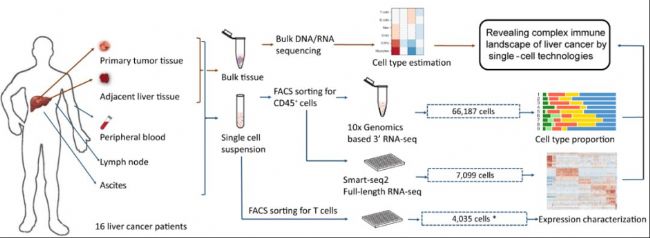
主要發現
- 不同組織的免疫組成有較大差異,腫瘤中的巨噬細胞構成腹水中髓系細胞的主要來源。
- 腫瘤中的巨噬細胞呈現兩種不同的狀態(TAM-like和MDSC-like)。
- 腫瘤中的LAMP3+ DC是成熟態的DC,具有向肝淋巴結遷移和與多種淋巴細胞相互作用的潛在能力。
參考文獻
1. Ziegenhain, C., et al., Comparative Analysis of Single-Cell RNA Sequencing Methods. Mol Cell, 2017. 65(4): p. 631-643 e4.
2. Vieth, B., et al., A systematic evaluation of single cell RNA-seq analysis pipelines. Nat Commun, 2019. 10(1): p. 4667.
3. Ramskold, D., et al., Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol, 2012. 30(8): p. 777-82.
4. Picelli, S., et al., Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods, 2013. 10(11): p. 1096-8.
5. Picelli, S., et al., Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc, 2014. 9(1): p. 171-81.
6. Bjorklund, A.K., et al., The heterogeneity of human CD127(+) innate lymphoid cells revealed by single-cell RNA sequencing. Nat Immunol, 2016. 17(4): p. 451-60.
7. Lambrechts, D., et al., Phenotype molding of stromal cells in the lung tumor microenvironment. Nat Med, 2018. 24(8): p. 1277-1289.
8. Zhang, Q., et al., Landscape and Dynamics of Single Immune Cells in Hepatocellular Carcinoma. Cell, 2019. 179(4): p. 829-845 e20.
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com