免疫系統與腫瘤細胞相互作用研究理論大解析
一、免疫-腫瘤互作的基礎機制
1. 免疫監視與腫瘤逃逸
正常情況下,免疫系統通過免疫監視機制識別并清除體內異常增生的細胞,包括潛在的惡性腫瘤細胞。該過程涉及固有免疫反應(如自然殺傷細胞、巨噬細胞等的直接殺傷)和適應性免疫反應(如T淋巴細胞、B淋巴細胞介導的特異性免疫應答)。然而,腫瘤細胞能夠通過多種策略逃避免疫系統的監控與清除,這些策略包括但不限于:
1. 免疫監視與腫瘤逃逸
正常情況下,免疫系統通過免疫監視機制識別并清除體內異常增生的細胞,包括潛在的惡性腫瘤細胞。該過程涉及固有免疫反應(如自然殺傷細胞、巨噬細胞等的直接殺傷)和適應性免疫反應(如T淋巴細胞、B淋巴細胞介導的特異性免疫應答)。然而,腫瘤細胞能夠通過多種策略逃避免疫系統的監控與清除,這些策略包括但不限于:
- 免疫抑制微環境的構建:腫瘤細胞分泌免疫抑制因子(如轉化生長因子TGF-β、白介素10IL-10、吲哚胺2, 3-雙加氧酶IDO等)、誘導免疫檢查點分子(如細胞程序性死亡-配體PD-L1、細胞毒性T淋巴細胞相關蛋白CTLA-4)表達,以及招募免疫抑制性細胞(如調節性T細胞、髓源性抑制細胞等),共同營造有利于腫瘤生長且抑制免疫活性的微環境。
- 抗原呈遞障礙:腫瘤細胞可能下調MHC分子表達,減少腫瘤抗原的呈現,從而降低被T細胞識別的概率。
- 免疫編輯:腫瘤細胞通過進化選擇,產生免疫逃逸的能力,使得免疫系統無法有效清除。
2. 循環腫瘤細胞與免疫細胞的相互作用
循環腫瘤細胞(CTCs)是存在于外周血中的各類腫瘤細胞的統稱,是從原發灶或轉移灶脫落進入血液循環的腫瘤細胞,它們與免疫細胞的相互作用對腫瘤的遠處播散至關重要[1]。研究表明,CTCs能夠通過與免疫細胞(如NK細胞、T細胞、巨噬細胞)的直接接觸或釋放可溶性因子,影響免疫細胞的功能,如抑制其殺傷活性、誘導免疫耐受或促進炎癥反應,有助于CTCs的存活與定植。
二、免疫-腫瘤互作的最新研究進展
1. 單細胞與空間轉錄組學技術的應用
高通量測序技術的進步,尤其是單細胞RNA測序(scRNA-seq)和空間轉錄組學,為深入解析腫瘤內復雜免疫微環境提供了前所未有的視角。這些技術揭示了腫瘤內部異質性、免疫細胞亞群的精細分型、細胞間通訊網絡以及動態變化過程,有助于闡明腫瘤免疫逃逸的具體機制和尋找新的免疫治療靶點。
2. 計算工具與算法的發展
例如,研究工作者在《Nature Methods》上發表的Cyto Community算法,這是一種針對單細胞空間圖譜進行無監督和有監督學習的計算工具,用于識別條件特異性的細胞鄰域協作,為解析腫瘤-免疫互作機制提供了有力的分析手段[2]。
3. 免疫治療新策略與藥物研發
針對免疫-腫瘤互作機制的深入理解,推動了新型免疫療法的研發,如免疫檢查點抑制劑(如PD-1/PD-L1、CTLA-4抑制劑)、CAR-T細胞療法、腫瘤疫苗、免疫激動劑等。此外,聯合療法也顯示出顯著的臨床效果,體現了精準調控免疫-腫瘤互作以實現高效抗癌治療的可能性。
三、JuLI™ Stage活細胞成像分析系統在免疫-腫瘤互作研究中的應用
奎克泰生物的JuLI™系列實時活細胞成像分析系統能夠為免疫-腫瘤互作研究提供所需要的工具。JuLI™系列設備操作簡單,無需復雜人工操作,省時省力;無需頻繁取出樣本觀察,提供穩定的生長環境;實時觀察,延時記錄,全面呈現免疫-腫瘤互作研究中細胞樣本的培養情況,獲取大量數據后進行分析。
JuLI™ Stage活細胞成像分析系統能夠放置于培養箱內,具有全自動X-Y-Z軸、三色熒光、自動/手動對焦、Z-Stack、圖像拼接等功能。可以對細胞樣本進行實時觀察,拍攝記錄生長周期的全過程,在保證樣本生長環境穩定的情況下,同時對樣本拍照,形成視頻,提供免疫-腫瘤互作實驗的量化結果。

圖1 JuLI™ Stage放置在細胞培養箱內工作
借助JuLI™ Stage進行免疫-腫瘤互作實驗,對癌細胞進行熒光標記 ,在GFP通道下觀察,免疫細胞在明場下觀察。對明場和GFP通道下樣本進行延時攝影,并自動合成視頻。下圖分別展示了在明場和GFP通道下圖像合并的結果(圖2)以及單獨GFP通道下T細胞成像結果(圖3)。

圖2 明場和GFP通道下圖像合并的結果

圖3 單獨GFP通道下癌細胞成像結果
拍攝結束后,利用JuLI™ Stage自帶的分析軟件,對視頻結果進行統一分析(圖4)。
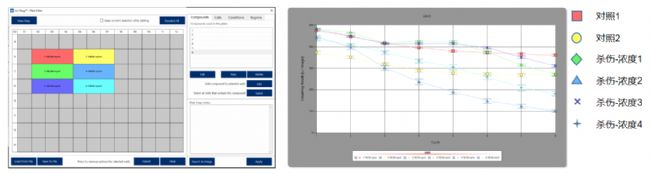
圖4 免疫細胞-癌細胞互作結果分析
參考文獻:
[1]Zhao QW, Situ B, Zheng L. [Current progress in research of circulating tumor cells]. Nan Fang Yi Ke Da Xue Xue Bao. 2017 Oct 20;37(10):1423-1426.
[2]Hu Y, Rong J, Xu Y, Xie R, Peng J, Gao L, Tan K. Unsupervised and supervised discovery of tissue cellular neighborhoods from cell phenotypes. Nat Methods. 2024 Feb;21(2):267-278.(IF48)
[1]Zhao QW, Situ B, Zheng L. [Current progress in research of circulating tumor cells]. Nan Fang Yi Ke Da Xue Xue Bao. 2017 Oct 20;37(10):1423-1426.
[2]Hu Y, Rong J, Xu Y, Xie R, Peng J, Gao L, Tan K. Unsupervised and supervised discovery of tissue cellular neighborhoods from cell phenotypes. Nat Methods. 2024 Feb;21(2):267-278.(IF48)
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com