Science文獻解讀:人類大腦的單細胞DNA甲基化和3D基因組結構
高通通量表觀基因組分析技術可用于闡明大腦中細胞復雜性的基因調控程序。5'-甲基胞嘧啶 (5mCs)是哺乳動物基因組中最常見的修飾堿基,大多數5mCs發生在胞嘧啶-鳥嘌呤二核苷酸(CpGs)上。CG差異甲基化區域 (DMRs)通常是順式調控元件(CREs)的標志。在脊椎動物的神經系統中,5mCs也在非CpG(或CH,H=A、C或T)中被大量檢測到。CG和CH甲基化(mCG和mCH)在大腦發育過程中高度動態變化,并表現出細胞類型特異性。mCG和mCH對于基因調控和大腦功能至關重要。因此基因調控需要適當的染色質折疊3D構象,這些構象被組織成活性(A)或抑制性(B)區、拓撲關聯域(TADs)和染色質環(chromatin loops)。這些3D結構促進了基因啟動子與其調控元件之間的互作,提供了額外但關鍵的調控機制層面。DNA甲基化與染色質構象互作,且過程高度相關。基于高通量表觀基因組分析技術對大腦細胞的表觀基因組表征分析可以加深對人類大腦的復雜性基因調控的理解。
2023年10月13日,索爾克生物研究所Joseph R. Ecker團隊聯合其他團隊研究人員在《科學》(SCIENCE)雜志上發表了“Single-cell DNA methylation and 3D genome architecture in the human brain”研究論文,通過使用單核表觀基因組測序技術,全面分析成年人類大腦皮層和亞皮層區域中的DNA甲基化和染色質構象,展示了人類大腦的單細胞DNA甲基化和3D基因組結構圖譜,闡明了整個大腦中細胞的細胞類型特異性和不同的表觀遺傳結構。

研究摘要:
闡明復雜細胞類型的基因調控程序對于理解健康和疾病中的大腦功能至關重要。本研究通過在3個成年男性大腦的46個區域對517k個細胞(399k神經元和118k非神經元)中以單細胞分辨率的DNA甲基化和染色質構象來全面描繪人腦細胞表觀基因組譜。研究共鑒定出188種細胞類型并表征其分子特征。綜合分析揭示了DNA甲基化、染色質可及性、染色質組織和跨細胞類型,皮質區域和基底神經節結構和基因表達在細胞類型、皮層區域和基底神經節結構中的一致變化。進一步開發了使用靶向基因組位點的甲基化狀態可靠預測腦細胞類型的scMCodes方法。這種多模式表觀基因組腦細胞圖譜為成人大腦中細胞類型特異性基因調控的復雜性提供了新的見解。
小編就本文中的DNA甲基化研究內容進行解讀分享。
甲基化研究思路
樣本:
517k 細胞(399k 神經元和 118k 非神經元),來自 3 個成年男性大腦的 46 個區域
技術:
- snmC-seq3(mC) 在單細胞水平上分析所有 46 個大腦區域的 DNA 甲基化 (DNAm)
- single-nucleus methylation sequencing單核亞硫酸鹽轉化的甲基胞嘧啶測序方法
- snm3C-seq(m3C) 檢測單細胞 DNA 甲基化和染色質構象
研究結果
(1)基于表觀基因組的腦細胞類型分類法
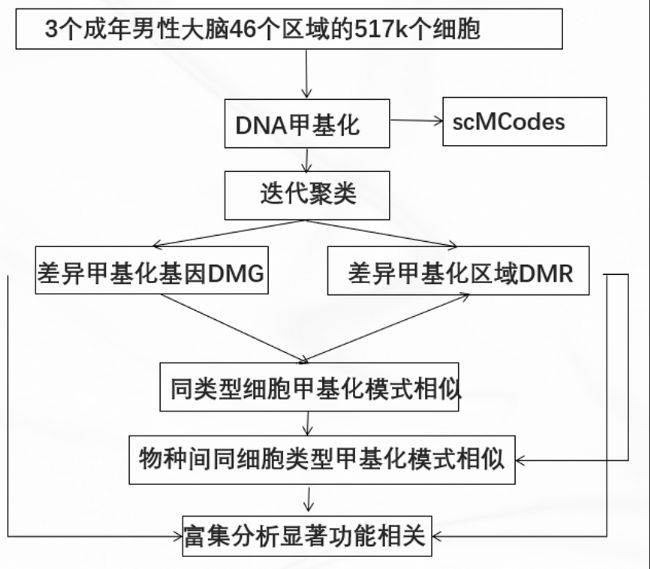
解剖了46個大腦區域,包括大腦皮層(CX,22個區域)、基底前腦(BF,2個區域)、基底核(BN,11個區域)、海馬體(HIP,5個區域)、丘腦(THM,2個區域)、中腦(MB,1個區域)、腦橋(PN,1個區域)和小腦(CB,2個區域)(圖1A)。大多數區域設置三個成年男性供體的三個生物學重復,兩個杏仁核區域除外(BM和CEN,各兩個重復)。熒光激活細胞核分選(FANS)用于分離每個樣品中90%的NeuN陽性細胞和10%的NeuN陰性細胞。然后使用snmC-seq3(“mC”)在單細胞水平上分析所有46個大腦區域的DNA甲基化(DNAm)。此外利用snm3C-seq(“m3C”)同時分析CX、BF和BN的17個大腦區域的單細胞DNA甲基化和染色質構象。經過嚴格質控,共378940 mC和145070 m3C細胞核用于進一步分析。每個mC細胞核平均產生0.94M過濾reads,而每個m3C核產生約2.2M reads和406k個染色質可及性。這樣的數據質量能夠可靠地分析各種基因組特征上的DNAm,鑒定可變甲基化區域,并精確定位不同大腦細胞類型的拓撲關聯域(TADs)和染色質環。

圖1:使用snmC-seq3和snm3C-seq技術對人類大腦細胞進行表觀基因組分析。
A. 人類大腦結構和區域覆蓋。
B. snmC-seq3和snm3C-seq的分析模式圖。
C. 人腦核的迭代聚類和注釋: 使用t-SNE技術對整個mC數據集、抑制性/非端腦神經元細胞類以及SubCtx-Cplx主要類型的細胞進行可視化,根據相應迭代中注釋的細胞組著色。
D. 主要類型的穩健樹狀圖和亞型數量、大腦結構和供體來源的meta信息。
E. SubCtx-Cplx主要類型的興奮性和抑制性標記(SLC17A1和GAD1)的CH甲基化水平。
F. 按解剖區域著色的人類大腦細胞。
G. snm3C-seq分析的人腦核的2D可視化。
H. 大腦細胞類型間整體CG-和CH甲基化變化。
I. 整體DNA甲基化與MECP2和DNMT1基因表達的相關性。
通過對mC數據集進行迭代聚類(iterative clustering),前腦分化出了最前端的端腦(telencephalon)和緊隨其后的間腦(diencephalon),后腦分裂出了后腦(metencephalon)和最末端的末腦(myelencephalon),中腦還是中腦,沒有進一步分裂。
細胞類型通過神經細胞的CH位點的低甲基化基因標記,和非神經細胞的CG位點的低甲基化標記區分(之前提到在脊椎動物神經元系統中,5mCs 在非 CG(或 CH,H=A、C 或 T)背景下也能被大量檢測到)
盡管在不同供體中某些細胞類型的比例略有不同,但所有major types和subtypes的分類都是一致的
樹枝圖顯示了主要類型和亞型之間的關系。端腦興奮性神經元和抑制性/非端腦神經元與非神經元細胞很好地區分開來,每種類型都形成了一個特定的支系,但 CB 和 PKJ 與非神經元細胞類型歸為一類,這可能是由于它們的整體 CG- 甲基化和 CH-甲基化程度相似。
分析結果表明:
- 378940 mC和145070 m3C細胞核被證實可檢測跨基因組特征的DNAm;
- 通過對mC數據集的迭代聚類,首先將細胞核分為端腦興奮性神經元、抑制性/非端腦神經元和非神經元細胞(40種主要類型和188種亞型);
- 根據神經元細胞的CH低甲基化基因標記物和非神經元細胞的CG低甲基化標記物對細胞類型進行注釋。
- 整體甲基化水平在主要類型之間有所差異:mCG為77.7%-85.5%,mCH為0.8%-10.7%。
- 非神經元和顆粒細胞(DG和CB)的主要類型在mCG和mCH的整體評分最低。
- 皮層抑制性神經元具有最高的mCG水平,而丘腦、中腦和腦橋的某些非端腦神經元表現出最高的mCH水平。
- 細胞類型的整體甲基化與DNA甲基化reader和修飾因子的基因表達相對應。
- 主要mCH reader MECP2表達與整體mCH呈正相關(皮爾遜相關系數,PCC=0.39),與mCG弱相關(PCC=0.17)。
- DNA甲基轉移酶DNMT1在其表達與mCG之間呈強正相關(PCC=0.63),且DNMT1表達與mCH相關性更高(PCC=0.72)。
- DNMT1與mCH之間可能存在某種尚未被發現的相關性。
(2)基因組組織與其他分子模式之間的關系
作者研究不同3D結構特征與其他表觀基因組模式(mCG、mCH和開放染色質)之間的相關性。研究結果揭示了在所有神經細胞類型中,mCG和mCH都與三維基因組組織呈負相關,但與染色質開放性存在正相關的關系,表明DNA甲基化可能促進CTCF和cohesin。
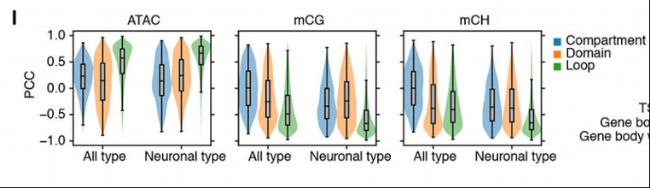
圖2-I:在所有基因(左)或僅最高差異表達基因(DEGs)(右)的所有主要類型中,區室得分、邊界概率或環互作強度與ATAC信號、bin的mCG和mCH分數之間的皮爾遜相關系數(PCC)。
在DNAm和三維基因組結構之間觀察到的負相關性可能是由于DNAm對驅動基因組折疊的因子(如CTCF)的結合產生了影響,通過高階結構的形成招募或排除了甲基化寫入因子或清除因子(如DNMTs和TETs),或者是甲基化和基因組組織的共同調控因子。
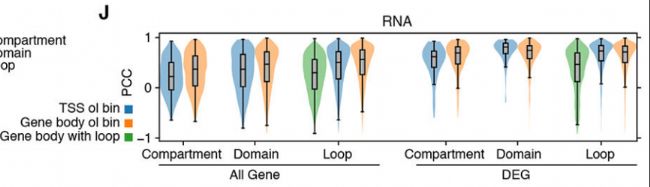
圖2-J:使用所有基因(左)或最高DEG(右)的所有主要類型中不同類別的重疊(x軸),隔室得分、邊界概率或環互作強度和基因表達之間的PCC。
結果表明:
- 基因表達也與三維(3D)基因組結構相關,特別是對于細胞類型特異性基因。
- 在所有神經元主要類型中,1099(1,358)個最高差異表達基因(DEGs)與其基因體或啟動子重疊的所有三個結構特征顯示出強烈的正相關性。基因表達和/或區塊結構信號的變異性增加與它們之間更高的正相關性相關聯,這證實了差異結構特征和差異基因表達之間的重疊。
- 這些(負)相關性表明,活性區、強結構域和環路相互作用以及開放染色質和甲基化耗竭之間存在協調關系,與活性染色質狀態相對應。
- 所有細胞類型之間的相關性普遍弱于神經元單獨存在的相關性。
(3)細胞類型特異性DNA甲基化模式和相關基因調控譜:CG和CH甲基化與染色質構象的整合揭示了不同的細胞類型調控動動態
為了描繪細胞類型特異性的甲基化譜,作者在188種大腦細胞亞型中鑒定了24455個CH型和13096個CG型差異甲基化基因(CG-DMGs)以及2059466個CG型差異甲基化區域(CG-DMRs)(圖3A)。除了為腦細胞身份提供獨特的表觀遺傳標記外,這些甲基化模式還提供了關鍵的見解,以理解腦細胞中的基因調控程序。基因體甲基化與基因表達呈負相關,DMRs作為潛在的順式調控元件(CREs),且轉錄因子(TF)motif暗示了候選的細胞類型特異性調控因子。
如果是低甲基化DMGs,且其motif在相同細胞類型的低甲基化DMRs(hypo-DMRs)中富集,就將TFs分配給特定的細胞類型。總共有612個TFs被分配到主要的神經元類型和亞型中,它們可能在塑造和維持細胞身份方面發揮重要角色。例如,TBR1被分配到深層興奮性神經元,特別是L6-CT和L6b(圖3B),并注意到它在皮質外向投射神經元的發育中起著決定命運的作用。ZNF423和EBF2都被分配到小腦細胞類型(圖3B)。兩者對小腦的發育至關重要,而EBF2尤其介導Purkinje細胞的遷移。
進一步分析亞型突出了TF利用的變化。例如TF PBX3,TF PBX3屬于紋狀體中普遍存在的MSN-D1主要類型,僅在紋狀體區室的亞型中被低甲基化,而在紋狀體的基質區室中沒有被低甲基化,這表明 PBX3 更傾向于在紋狀體中表達,印證了之前的觀察結果。對 PBX3 的潛在結合位點(具有 PBX3 motif的hypo-DMRs)的進一步研究表明,紋狀體亞型的平均甲基化分數較低,表明這個TF在紋狀體中具有特定室的調控作用。
整合差異甲基化基因(DMGs)、差異甲基化區域(DMRs)和差異環(differential loops)來精確定位每種細胞類型的潛在順式調控元件(CREs)(圖3C)。如果一個基因的轉錄起始位點(TSS)位于DMR的5 Mb范圍內,那么這個基因就被認為是與DMR相關聯。進一步細化保留與環或差異環(DL)的兩個錨點重疊的DMR-DMG對。計算不同細胞亞型中DMR的mCG分數與基因體的mCH分數之間的皮爾遜相關系數(PCC)來評估這種關聯。特別對于經過DL篩選的DMRs,觀察到了增強的相關性(圖3D),這些DMRs也顯示出與開放染色質區域的重疊增加(圖3E)。在1122919個DMRs和12327個基因之間鑒定出3.2M潛在的調控性DMR/基因對。這些DMRs、DMGs的甲基化分數以及它們互作強度(環)呈現出(負)相關性(圖3F),這些因素共同協調特定的基因調控程序。例如,編碼突觸結合蛋白-1(Synaptotagmin-1)的基因SYT1,在L2/3-IT神經元中展現出較低的遠端DMRs和SYT1基因體的甲基化分數,并且與DMRs和啟動子之間的相互作用比MSN-D1神經元更強(圖3G),導致SYT1在L2/3-IT中的表達高于MSN-D1(圖3G)。總體而言,CG和CH甲基化與染色質構象的整合揭示了不同細胞類型的調控動態。
通過全基因組關聯研究(GWAS)確定了許多與腦部疾病相關的非編碼位點,其中很多位于增強子區域。DMRs和loops有助于將這些遺傳變異定位到特定細胞類型的調控元件上。使用連鎖不平衡得分回歸(LDSC)方法,檢測到20種腦部疾病或性狀與人類大腦細胞中的DMRs或與環重疊的DMRs之間的關聯(圖3H)。精神分裂癥、雙相情感障礙和神經質風險變異在皮質和海馬體的興奮性神經元的低甲基化DMRs中顯著富集,而阿爾茨海默病(AD)與小膠質細胞(MGC;圖3H)相關。煙草使用障礙變異與基底節的Foxp2細胞類型相關(圖3H),這是一個與煙草成癮相關的區域。對疾病風險變異的進一步探索揭示了對基因調控的多樣化影響。盡管許多細胞類型與相同的疾病有關,但它們所涉及的風險變異可能多樣。例如,精神分裂癥風險變異rs2789588在L2/3-IT和L6-CT神經元中都有所涉及,具有相似的表觀遺傳特征,但rs17194490僅在L2/3-IT中涉及,與相應基因的特定DNA低甲基化、更強的長距離相互作用和與L6-CT相比更高的基因表達相關。

圖3:大腦細胞中的基因調控
(4)人、鼠腦細胞類型和DMR的保守性
研究人員分析了靈長類動物(人類)和嚙齒類動物(小鼠)之間在大腦細胞類型上的保守性。通過比較人類和小鼠的單核DNA甲基化圖譜,范圍覆蓋大腦皮層、基底前腦、基底核和海馬體等相應區域。整合分析表明,人類大腦中定義的三種主要類型與小鼠腦細胞不一致(圖5A),小鼠L4-IT神經元只對應于人類L4-IT神經元亞群(圖5B),證實了人類L4-IT神經元中存在更大的異質性。人類海馬體中的HIP-Misc1神經元與一些小鼠皮層IT神經元整合,而HIP-Misc2神經元與任何小鼠細胞類型均不對應。平行snRNA數據集驗證了這兩種人類海馬體細胞類型(圖5C)。盡管未匹配的細胞類型需要進一步研究,但主要類型的分類在人類和小鼠之間更廣泛的大腦區域通常是保守的(圖5A),而對于相應的細胞類型,人類的整體CG和CH甲基化水平始終高于小鼠(圖5D)。
為了比較人腦和小鼠大腦之間的基因調控,研究者使用liftOver匹配在單個物種內鑒定的主要類型低甲基化差異區域(hypo-DMRs)(圖5E)。跨細胞類型的40~60%的hypo-DMRs在另一種物種中具有同源序列(將這些DMRs稱為OrthSeqs)。大約一半的OrthSeqs在另一種物種中的同源物也是hypo-DMRs(OrthDMRs)。大多數(95%)OrthDMRs相互匹配(CnsvDMRs;圖5F)。CnsvDMRs的甲基化分數在人鼠細胞類型間顯示出顯著的相關性(圖5, G和H),表明功能在物種間保守。
研究者進一步選擇了相關性最高的DMRs(hcCnsvDMRs,圖5G)。hcCnsvDMRs的功能富集分析表明,它們在與前腦發育相關的生物過程和與樹突和突觸相關的細胞組分中富集(圖5F和G)。與小鼠前腦的組蛋白修飾比較表明,這些DMRs在異染質區域(H3K9me3)中缺失,在增強子(H3K27ac和H3K4me1)、啟動子(H3K4me3)和poised增強子(H3K27me3)區域中富集。根據染色質可及性將hcCnsvDMRs進一步分類為開放或關閉狀態,結果表明開放DMRs在增強子和啟動子中富集。而關閉的DMRs在poised增強子中特別富集(圖5I),這些增強子可能在發育過程中活躍。
物種間的甲基化保守性暗示了通過比較表觀遺傳學發現增強子的策略。例如,Pvalb神經元的特定基因INPP5J,有許多遠端和近端的hcCnsvDMRs與匹配的染色質可及區域重疊(圖5J),包括兩個被驗證為小鼠Pvalb神經元病毒靶向的特定增強子(圖5J)。
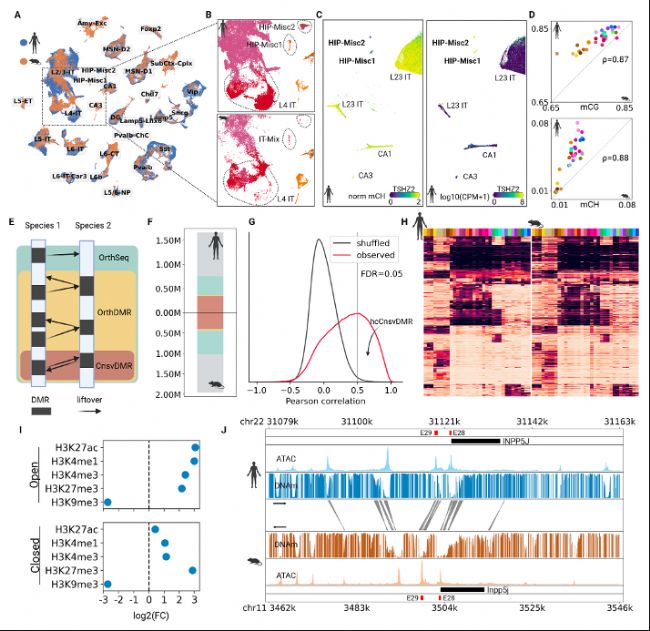
圖5:人類和小鼠大腦細胞甲基化組的跨物種比較。
A. 2D t-SNE可視化人類和小鼠大腦之間的單細胞甲基組化整合。
B. L4-IT、HIP-Misc1和HIP-Misc2細胞類型中,人類和小鼠大腦的細胞類型差異。
C. TF TSHZ2在HIP-Misc1和HIP-Misc2細胞類型中,CH低甲基化和基因表達。
D. 人類和小鼠之間保守細胞類型的整體mCH和mCG相關性。
E. 跨物種匹配細胞類型DMRs示意圖。
F. 大約50%的DMRs在另一個物種中具有同源序列,其中約25%是相互對應的DMRs。
G. 跨物種DMR甲基化相關性分布(紅色)和隨機背景(黑色)。
H. hcCnsvDMRs的甲基化分數示例。
I. hcCnsvDMRs在組蛋白修飾標記中的富集情況。
J. 在Pvalb主要類型中,圍繞INPP5J基因的hcCnsvDMRs的瀏覽器視圖。
(5)單細胞甲基化條形碼 (scMCodes) 可靠地預測人腦細胞身份。
細胞基因組中的DNA甲基化變化包含了代表過去和現在基因調控事件的分子“印記”,許多CpG位點上大腦細胞類型的DNA甲基化模式具有高度特異性。為此,作者設計了單細胞甲基化條碼(scMCodes),通過選定CpG位點的甲基化狀態來確定單個細胞水平上的大腦細胞類型(圖6A)。
首先通過迭代選擇區分大腦細胞類型的CpG位點,然后根據其在不同細胞類型間的甲基化模式,將這些位點進一步聚類成39k個群體。接著通過機器學習模型交叉驗證評估了它們的細胞類型預測能力,最終選擇800個群體共12000個CpG位點作為scMCodes(圖6B和C),以在保持特征數最小化同時實現良好的預測能力。這些scMCodes達到約93%的準確率(圖6D)。
在本研究的三個供體和一個外部個體之間進行了跨供體測試。結果顯示高預測準確率(92~96%;圖6E),證明了scMCode方法在跨個體上的穩健性。單細胞測序的基因組覆蓋有限,平均每個細胞中只檢測到約200個scMCodes的CpG位點(圖6F),強調了scMCode使用少數選定的甲基化位點來確定人類大腦細胞類型的scMCode方法有效性。

圖6:大腦細胞類型的snMCodes。
A. snMCodes的工作流程。
B. 來自所有三位供體的snMCodes。
C. snMCode特征的細胞類型特異性示例。
D. snMCodes在預測細胞類型時的混淆矩陣熱圖。
E. 跨供體測試中的細胞類型預測準確率。
F. snMCodes能夠在單細胞分辨率下使用有限數量的CpG位點預測人類細胞類型
Discussion
研究人員編制了一個全面的單細胞DNA甲基化和人類大腦3D基因組結構圖譜,并比較了不同大腦區域相同細胞類型的表觀遺傳多樣性。DNA甲基化(DNAm)中編碼的復雜調控信息使我們能夠提煉出一組單細胞甲基化條碼(scMCodes),用于可靠的細胞類型識別。鑒于循環游離DNA(cfDNA)甲基化已被認定為癌癥診斷的有力工具,并且為腦部疾病提供了有希望的生物標志物。
研究通過單細胞分析技術揭示的DNA甲基化模式在理解和分類大腦細胞類型中的重要性。研究人員利用這些信息開發了scMCodes,可以在單細胞水平上準確地鑒定和分類細胞類型。此外,cfDNA中的甲基化模式的研究不僅在癌癥診斷中顯示出潛力,也為腦部疾病的生物標志物發現提供了新的方向。這些發現可能對開發新的診斷方法和治療策略具有重要意義。
參考文獻:
Tian W, Zhou J, Bartlett A, Zeng Q, Liu H, Castanon RG, Kenworthy M, Altshul J, Valadon C, Aldridge A, Nery JR, Chen H, Xu J, Johnson ND, Lucero J, Osteen JK, Emerson N, Rink J, Lee J, Li YE, Siletti K, Liem M, Claffey N, O'Connor C, Yanny AM, Nyhus J, Dee N, Casper T, Shapovalova N, Hirschstein D, Ding SL, Hodge R, Levi BP, Keene CD, Linnarsson S, Lein E, Ren B, Behrens MM, Ecker JR. Single-cell DNA methylation and 3D genome architecture in the human brain. Science. 2023 Oct 13;382(6667):eadf5357. doi: 10.1126/science.adf5357. PubMed PMID: 37824674.