全基因組cfDNA甲基化分析在提高早期乳腺癌無創診斷成像準確性的應用
乳腺癌早期發現是提高患者預后和生存率的關鍵。基于乳房X線片和超聲技術的乳腺影像報告和數據系統(BI-RADS)被廣泛用于乳腺癌的早期診斷,但這種方法的假陽性率較高,會導致不必要的活檢,尤其是對于BI-RADS-4類的患者來說。而攜帶DNA甲基化信息的游離DNA(cfDNA)已經成為一種非侵入性的癌癥檢測方法。在本文中,作者進行了一項前瞻性多中心研究,利用全基因組重亞硫酸鹽測序數據來研究203名疑似惡性乳腺病變的女性患者cfDNA甲基化標志物的臨床應用。cfDNA主要富集在低甲基化的基因組區域。作者設計了一套方法來挖掘最佳的DNA甲基化標記物,顯著提高了BI-RADS 4類患者的早期診斷率(AUC從0.78-0.79到0.93-0.94)。作者通過開展基于血液的全基因組DNA甲基化研究,以單堿基分辨率從良性腫瘤中檢測早期乳腺癌,結果表明結合液體活檢和傳統的成像診斷可以通過降低假陽性率和避免不必要的損害來改進當前乳腺癌早期診斷的臨床實踐。

方案設計
本研究收集了接受乳腺X線片和超聲檢查后活檢的210例BI-RADS 4類乳腺病變女性患者。從CHCAMS的活檢患者中收集了20個腫瘤樣本(10個惡性和10個良性),用于全基因組亞硫酸氫鹽測序(WGBS)。后續具體的研究方案如圖1所示。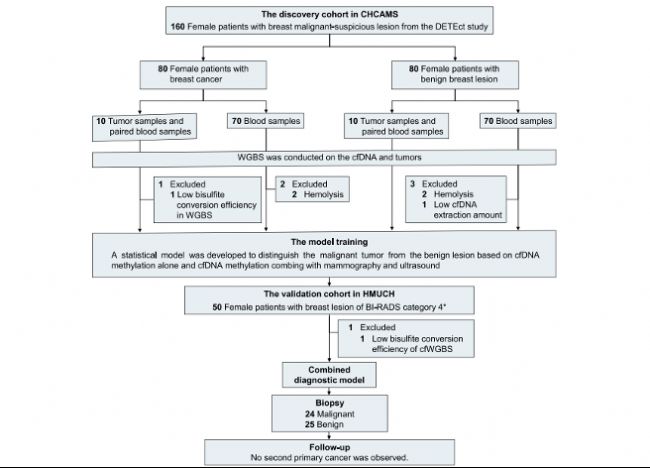 圖1
圖1
主要結果
作者利用WGBS檢測了每個受試者的cfDNA甲基化組。由于ctDNA在總cfDNA中的含量較低,作者設計了一個計算框架來提高cfDNA甲基化標志物的檢測(圖2),該框架包括基于腫瘤樣本的差異甲基化區域的識別、cfDNA富集分析、片段大小選擇、基于片段的cfDNA惡性比率的統計推斷以及早期乳腺癌預測模型的建立。
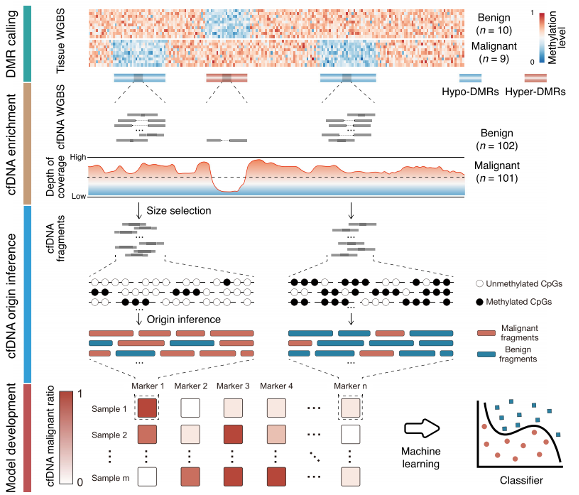
cfDNA甲基化標記的鑒定。研究發現WGBS的cfDNA片段在編碼區和基因間隔區富集,而在基因啟動子區域缺失(圖3A)。不同基因組區域的相應cfDNA數量與CpG密度和GC含量呈負相關(圖3B)。在9個惡性腫瘤和10個良性腫瘤樣本之間共鑒定出57,575個DMR(51,962個低DMR和5613個高DMR)。正如預期的那樣,高DMR患者的CpG密度顯著高于低DMR患者(p<0.0001;圖3C)。因此,在所有惡性和良性樣本中,低DMR中cfDNA片段的平均數量顯著高于高DMR中的cfDNA片段平均數量(p<0.0001;圖3D)。CpG富集的高DMRs中cfDNA片段的缺失可能是由于cfDNA中開放的染色質區域優先被消化。這些發現有助于作者優化低甲基化區域的cfDNA甲基化標記。異常低甲基化,一般在cfDNA豐富的基因體或基因間區的封閉染色質中,是各種惡性腫瘤的共同特征。為了保證高質量的cfDNA定量,本文選擇了低DMRs作為候選的DNA甲基化標記。
計算每個患者樣本的cfDNA惡性比率。利用發現隊列數據,篩選出10個區分惡性和良性血漿樣本的DMR。它們大多位于基因間區,其中惡性腫瘤患者的惡性比例明顯高于良性病變患者( p < 0.05 ;圖 3E)。但在10個甲基化標記中,有4個功能基因( RYR2、 RYR3、 GABRB3和 DCDC2C )和2個lncRNA ( AC096570.1和LINC00923 )。
基于cfDNA甲基化的早期乳腺癌預測模型。利用cfDNA分子標記物的惡性比率,使用隨機森林分類器(Random Forest)計算發現隊列中每個血漿樣本的cfDNA甲基化預測評分( cfMeth評分)。發現組和驗證組模型的曲線下面積( AUC )分別為0.89 (95 % CI , 0.84 ~ 0.94 ;圖 3F)和0.81 ( 95 % CI , 0.69 - 0.93 ;圖 3G ),優于BI-RADS結果(AUC = 0.78 - 0.79 )或相關腫瘤標志物檢測結果( CA15 - 3和CEA AUC = 0.57 - 0.70)。
cf Meth評分與影像診斷的組合模型。采用嶺回歸(ridge regression)方法在發現隊列中建立cf Meth評分與乳腺X光、超聲檢查的組合診斷模型。聯合模型的表現優于任何一種單獨的方法( AUC = 0.94,95 % CI,0.90-0.97 ;圖3H )。惡性組和良性組在發現隊列和驗證隊列中的組合評分分布均有統計學差異。聯合模型的截斷點被選為在發現隊列中假陰性率小于2 %,其敏感性為98.7 %,特異性為68.8 %,準確性為83.8 %。在驗證隊列中,性能相似( AUC = 0.93 [ 95 % CI , 0.84-1.00 ] );圖3I ),其敏感性為91.7 %,特異性為88.0 %,準確性為89.8 %,沒有過度擬合的證據。總體而言,聯合模型的檢出率分別為93.3 % ( 42 / 45 )、100 % ( 34 / 34 )、100 % ( 22 / 22 ),腫瘤Ⅰ、Ⅱ、Ⅲ期診斷的特異性為73.5 %。

圖3
結論
在單堿基分辨率下,作者進行了基于血液的全基因組DNA甲基化檢測早期乳腺癌的研究,結果顯示液體活檢與傳統的診斷顯像相結合可以提高早期乳腺癌診斷的準確性。

方案設計
本研究收集了接受乳腺X線片和超聲檢查后活檢的210例BI-RADS 4類乳腺病變女性患者。從CHCAMS的活檢患者中收集了20個腫瘤樣本(10個惡性和10個良性),用于全基因組亞硫酸氫鹽測序(WGBS)。后續具體的研究方案如圖1所示。
 圖1
圖1主要結果
作者利用WGBS檢測了每個受試者的cfDNA甲基化組。由于ctDNA在總cfDNA中的含量較低,作者設計了一個計算框架來提高cfDNA甲基化標志物的檢測(圖2),該框架包括基于腫瘤樣本的差異甲基化區域的識別、cfDNA富集分析、片段大小選擇、基于片段的cfDNA惡性比率的統計推斷以及早期乳腺癌預測模型的建立。

圖2
在文庫制備和測序后,作者通過分析WGBS數據(n=101例惡性和102例良性)預測了cfDNA長度的分布情況,結果顯示cfDNA長度分布呈現典型模式,在167bp處有顯著富集。cfDNA與19個組織樣本一起產生了總計4.0TB的WGBS數據,平均覆蓋參考基因組的88%,平均深度為11.2 x。cfDNA甲基化標記的鑒定。研究發現WGBS的cfDNA片段在編碼區和基因間隔區富集,而在基因啟動子區域缺失(圖3A)。不同基因組區域的相應cfDNA數量與CpG密度和GC含量呈負相關(圖3B)。在9個惡性腫瘤和10個良性腫瘤樣本之間共鑒定出57,575個DMR(51,962個低DMR和5613個高DMR)。正如預期的那樣,高DMR患者的CpG密度顯著高于低DMR患者(p<0.0001;圖3C)。因此,在所有惡性和良性樣本中,低DMR中cfDNA片段的平均數量顯著高于高DMR中的cfDNA片段平均數量(p<0.0001;圖3D)。CpG富集的高DMRs中cfDNA片段的缺失可能是由于cfDNA中開放的染色質區域優先被消化。這些發現有助于作者優化低甲基化區域的cfDNA甲基化標記。異常低甲基化,一般在cfDNA豐富的基因體或基因間區的封閉染色質中,是各種惡性腫瘤的共同特征。為了保證高質量的cfDNA定量,本文選擇了低DMRs作為候選的DNA甲基化標記。
計算每個患者樣本的cfDNA惡性比率。利用發現隊列數據,篩選出10個區分惡性和良性血漿樣本的DMR。它們大多位于基因間區,其中惡性腫瘤患者的惡性比例明顯高于良性病變患者( p < 0.05 ;圖 3E)。但在10個甲基化標記中,有4個功能基因( RYR2、 RYR3、 GABRB3和 DCDC2C )和2個lncRNA ( AC096570.1和LINC00923 )。
基于cfDNA甲基化的早期乳腺癌預測模型。利用cfDNA分子標記物的惡性比率,使用隨機森林分類器(Random Forest)計算發現隊列中每個血漿樣本的cfDNA甲基化預測評分( cfMeth評分)。發現組和驗證組模型的曲線下面積( AUC )分別為0.89 (95 % CI , 0.84 ~ 0.94 ;圖 3F)和0.81 ( 95 % CI , 0.69 - 0.93 ;圖 3G ),優于BI-RADS結果(AUC = 0.78 - 0.79 )或相關腫瘤標志物檢測結果( CA15 - 3和CEA AUC = 0.57 - 0.70)。
cf Meth評分與影像診斷的組合模型。采用嶺回歸(ridge regression)方法在發現隊列中建立cf Meth評分與乳腺X光、超聲檢查的組合診斷模型。聯合模型的表現優于任何一種單獨的方法( AUC = 0.94,95 % CI,0.90-0.97 ;圖3H )。惡性組和良性組在發現隊列和驗證隊列中的組合評分分布均有統計學差異。聯合模型的截斷點被選為在發現隊列中假陰性率小于2 %,其敏感性為98.7 %,特異性為68.8 %,準確性為83.8 %。在驗證隊列中,性能相似( AUC = 0.93 [ 95 % CI , 0.84-1.00 ] );圖3I ),其敏感性為91.7 %,特異性為88.0 %,準確性為89.8 %,沒有過度擬合的證據。總體而言,聯合模型的檢出率分別為93.3 % ( 42 / 45 )、100 % ( 34 / 34 )、100 % ( 22 / 22 ),腫瘤Ⅰ、Ⅱ、Ⅲ期診斷的特異性為73.5 %。

圖3
在單堿基分辨率下,作者進行了基于血液的全基因組DNA甲基化檢測早期乳腺癌的研究,結果顯示液體活檢與傳統的診斷顯像相結合可以提高早期乳腺癌診斷的準確性。
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com