基于納米孔測序技術分析靶向STR檢測方法
1月18日,齊碳生物信息團隊獨立自主開發的NanoSTR,一種基于納米孔測序技術的靶向STR檢測方法在線發表于Frontiers in Molecular Biosciences (影響因子6.113);
 該方法同時拿到了國家發明專利的授權證書以及軟件著作權,再次實現了“產學研”的整體閉環,擴展了納米孔測序數據的分析手段,為推動納米孔測序技術在科學研究和臨床實踐中的應用提供了幫助。
該方法同時拿到了國家發明專利的授權證書以及軟件著作權,再次實現了“產學研”的整體閉環,擴展了納米孔測序數據的分析手段,為推動納米孔測序技術在科學研究和臨床實踐中的應用提供了幫助。

背景
短串聯重復序列(STR)廣泛存在于人類基因組中。研究已經證實了STR與30多種疾病有關,它們也常被用于法醫鑒定和親子鑒定等領域。
然而,由于納米孔測序原理和測序數據的特征問題帶來的挑戰,目前,基于納米孔測序進行STR檢測的方法還比較少。
因此,齊碳科技生信團隊開發了靶向STR檢測方法——NanoSTR (Github鏈接: https://github.com/langjidong/NanoSTR),該方法利用“多采樣”等統計學方法以及基于測序數據的Length-Number-Rank (LNR)信息進行檢測,相關原理如如圖1所示。
圖1. NanoSTR分析方法的原理圖。
成果概述
NanoSTR可用于基于納米孔測序的長讀長數據的STR檢測和基因分型,并且與現有的一些分析方法(例如Tandem-genotypes和TRiCoLOR)相比,其準確性和效率均有所提高,表現出了較高性能及一定的魯棒性。
NanoSTR使用無錯誤模擬數據顯示出與預期基因型100%的一致性,使用MinION和Qnome-3841測序平臺的標準品樣本(包含常染色體和Y染色體基因座),一致性可分別達>85%和>71%(如圖2、圖3所示)。雖然NanoSTR還需要進一步的優化和開發,但驗證數據表明:它作為通過納米孔測序檢測STR基因座的分析方法是有用的。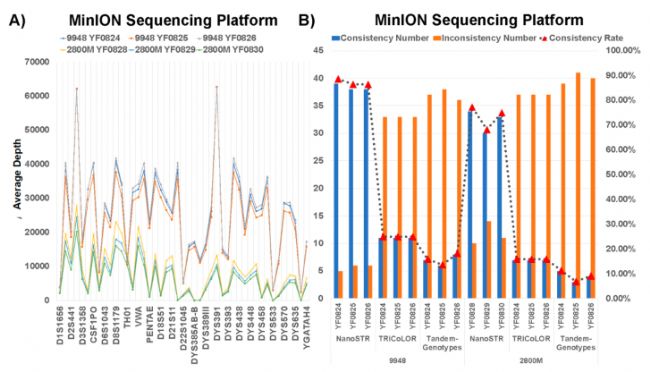
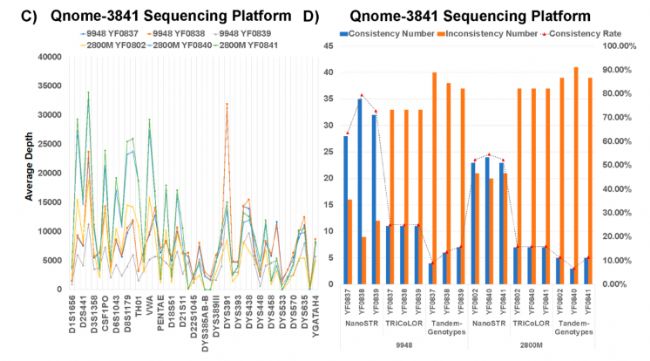 圖2. A) 6例標準品數據(MinION測序平臺)在各個靶向STR區間的平均深度分布圖;B) NanoSTR、TRiCoLOR和Tandem-Genotypes在各個標準品數據(MinION測序平臺)上的性能分析;C) 6例標準品數據(Qnome-3841測序平臺)在各個靶向STR區間的平均深度分布圖;B) NanoSTR、TRiCoLOR和Tandem-Genotypes在各個標準品數據(Qnome-3841測序平臺)上的性能分析。其中柱狀圖代表各個標準品與預期結果的分型一致個數(藍色)及不一致的個數(橙色),虛線連接的三角形符號代表與預期結果的一致率。
圖2. A) 6例標準品數據(MinION測序平臺)在各個靶向STR區間的平均深度分布圖;B) NanoSTR、TRiCoLOR和Tandem-Genotypes在各個標準品數據(MinION測序平臺)上的性能分析;C) 6例標準品數據(Qnome-3841測序平臺)在各個靶向STR區間的平均深度分布圖;B) NanoSTR、TRiCoLOR和Tandem-Genotypes在各個標準品數據(Qnome-3841測序平臺)上的性能分析。其中柱狀圖代表各個標準品與預期結果的分型一致個數(藍色)及不一致的個數(橙色),虛線連接的三角形符號代表與預期結果的一致率。
NanoSTR不僅一定程度上規避了納米孔測序數據特征造成的分型錯誤或無法分型的問題,而且無需建立基因組背景數據庫或將測序數據比對到人類基因組上,降低了計算資源的消耗,也不需要進行二次處理例如作圖來進行輔助或主觀判斷STR型別,節省了大量的分析時間。
結語
齊碳希望NanoSTR在豐富了基于納米孔測序檢測靶向STR分析工具的同時,還能拓寬納米孔測序技術在科學研究和臨床場景中的應用。
未來,齊碳生信團隊將聯合實驗技術團隊繼續開發STR檢測的新方法及優化NanoSTR的性能,進一步提高STR檢測的準確性,并且拓展更多其在科研和臨床實踐中的應用場景。
參考文獻:
[1]Lang J, Xu Z, Wang Y, Sun J, Yang Z. NanoSTR: A method for detection of target short tandem repeats based on nanopore sequencing data. Front Mol Biosci. 2023 Jan 18;10:1093519. doi: 10.3389/fmolb.2023.1093519. PMID: 36743210; PMCID: PMC9889824.


背景
短串聯重復序列(STR)廣泛存在于人類基因組中。研究已經證實了STR與30多種疾病有關,它們也常被用于法醫鑒定和親子鑒定等領域。
然而,由于納米孔測序原理和測序數據的特征問題帶來的挑戰,目前,基于納米孔測序進行STR檢測的方法還比較少。
因此,齊碳科技生信團隊開發了靶向STR檢測方法——NanoSTR (Github鏈接: https://github.com/langjidong/NanoSTR),該方法利用“多采樣”等統計學方法以及基于測序數據的Length-Number-Rank (LNR)信息進行檢測,相關原理如如圖1所示。

圖1. NanoSTR分析方法的原理圖。
成果概述
NanoSTR可用于基于納米孔測序的長讀長數據的STR檢測和基因分型,并且與現有的一些分析方法(例如Tandem-genotypes和TRiCoLOR)相比,其準確性和效率均有所提高,表現出了較高性能及一定的魯棒性。
NanoSTR使用無錯誤模擬數據顯示出與預期基因型100%的一致性,使用MinION和Qnome-3841測序平臺的標準品樣本(包含常染色體和Y染色體基因座),一致性可分別達>85%和>71%(如圖2、圖3所示)。雖然NanoSTR還需要進一步的優化和開發,但驗證數據表明:它作為通過納米孔測序檢測STR基因座的分析方法是有用的。


NanoSTR不僅一定程度上規避了納米孔測序數據特征造成的分型錯誤或無法分型的問題,而且無需建立基因組背景數據庫或將測序數據比對到人類基因組上,降低了計算資源的消耗,也不需要進行二次處理例如作圖來進行輔助或主觀判斷STR型別,節省了大量的分析時間。
結語
齊碳希望NanoSTR在豐富了基于納米孔測序檢測靶向STR分析工具的同時,還能拓寬納米孔測序技術在科學研究和臨床場景中的應用。
未來,齊碳生信團隊將聯合實驗技術團隊繼續開發STR檢測的新方法及優化NanoSTR的性能,進一步提高STR檢測的準確性,并且拓展更多其在科研和臨床實踐中的應用場景。
參考文獻:
[1]Lang J, Xu Z, Wang Y, Sun J, Yang Z. NanoSTR: A method for detection of target short tandem repeats based on nanopore sequencing data. Front Mol Biosci. 2023 Jan 18;10:1093519. doi: 10.3389/fmolb.2023.1093519. PMID: 36743210; PMCID: PMC9889824.
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com