超分辨率顯微技術在研究細胞結構中的應用和原理
STORM的數字圖像處理系統
“數字圖像處理是通過圖像數據進行相關功能處理和分析的技術。在storm(stochastic optical reconstruction microscopy)系統中,生物樣本信息經由光路放大后,投射到系統的主相機感光板上,轉化為數字圖像數據,由數字圖像處理系統統一進行處理。本文就storm的數字圖像處理系統給大家做個介紹。”
01成像原理
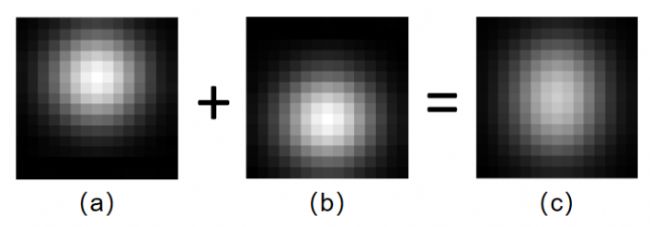
圖1光斑疊加
“無論一個樣品結構有多復雜,總是能夠描述為是由一個個點構成的。”這就是storm的核心原理,如圖2所示,假設圖1中的兩個光斑結構在不同時間分別進行閃爍,對閃爍的每一幀圖像數據中的高斯光斑進行捕捉和擬合,獲得高斯光斑的中心位置并記錄,在完成拍攝后,將所有記錄下的光斑信息進行疊加重構,便可獲得完整的結構信息。

圖2隨機光斑疊加
在storm系統中,通過生物試劑以及光學激發,利用計算機處理相機拍攝到的隨機熒光閃爍,并對閃爍的光斑進行統一分析,就可獲得生物樣本200納米以下的結構信息,如圖3為數字圖像處理系統處理后得到的細胞內部結構圖。
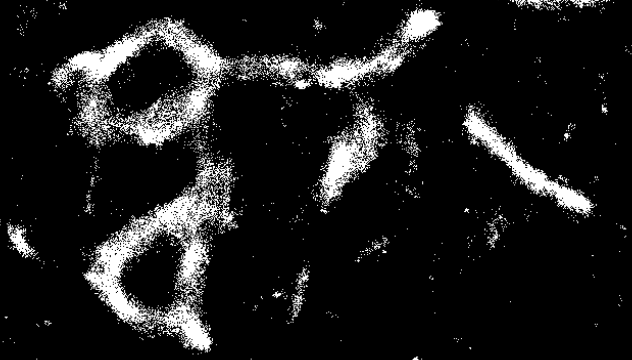
圖3storm拍攝細胞內部結構
02誤差矯正
串擾問題可以定義為熒光從一個通道到其他通道的泄漏,串擾的原因是因為多種熒光染料的發射光譜重疊。然而,二向色鏡無法完全阻擋重疊光譜,這就導致當前通道上會出現不屬于該通道的圖像信息。
圖4圖像中存在的串色
要解決串擾問題,首先要確定串擾的比例,拍攝單一染料的寬視場圖像,然后得到每個樣品中每個通道的平均強度,以形成包含所有九個平均強度的強度矩陣,通過圖像系統中的串色抑制算法,便可在原始圖像數據上做抑制,通過圖像數據計算的方式解決串色問題。
2、通道對齊
storm系統支持多通道同時成像,在硬件結構上,光路結構的差異和色差造成各個通道之間存在一定的誤差。
如圖6所示,紅通道(561nm激光)與綠通道(647nm激光)在拍攝金納米粒子時產生的圖像分離。

圖5通道的偏移
經實驗發現,由于光路傳遞的原因,會造成每個通道會產生不同的程度的畸變與距離偏差,為了抵消這些系統誤差,數字圖像系統利用金納米粒子對系統誤差進行修正,在拍攝好金納米粒子的寬場圖像后,對每個熒光點在x與y方向進行通道誤差計算,獲得每個金納米點與對應通道上的最近鄰匹配點的誤差數據,通過對齊算法,最終確定當前通道與對應通道之間的誤差關系。
3、3D校準
storm不僅可以拍攝生物樣本的二維信息,還可以對生物樣本的三維信息進行分析與重構,這時就需要對三維方向上的誤差進行校準,以便展示給用戶的是最準確的信息。
三維信息獲取的一種方式是依靠系統光路中的柱面鏡對成像造成的影響,如圖6所示在加入柱面鏡后,當發光點位于焦平面下方時,捕獲到的光斑為豎直方向拉伸,同理,當發光點位于焦平面上方時,捕獲到的光斑為橫方向拉伸。利用這個特性,通過對光斑形態的分析,便可獲得每個光斑在超高亞像素上的長寬邊界差值與深度信息的曲線關系,從而反向推導出該發光點的實際深度信息。

圖6柱面鏡對光斑的影響
在獲得每個閃爍光斑的深度信息后,storm便可對拍攝樣本進行3D重構,獲得如下效果:

圖7重構后的肌動蛋白長絲的 x-y 和 x-z 橫截面
03 總結
參考文獻
[1] 張世超, 李思黽, 楊光,等. 3D-STORM超分辨成像中單分子軸向定位精度優化研究[J]. 光子學報, 2015(10):6.
[2] Rust M J . Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM).[J]. Nature Methods, 2010, 3.
[3] Yu, Lujia. Three color super-resolution localization microscopy for optical mapping of stretched DNAs in nanochannels.Thesis (M.Phil.)--Hong Kong University of Science and Technology, 2019
標簽:
超分辨率顯微技術
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com