3月單細胞轉錄組重要研究文獻匯總
1.單細胞測序用于生殖領域研究持續火熱
2.單細胞測序用于免疫研究多點開花
(1)Memory CD4+ T cells研究
(3)干細胞記憶樣T細胞的產生過程追蹤

4.單細胞轉錄組測序在植物領域嶄露頭角
相比動物的單細胞研究,由于植物具有細胞壁的特點,使得植物的單細胞研究相比較難,但是繼2月份的擬南芥根組織單細胞轉錄組學研究[8],本月在《Developmental Cell》上又發表了一篇基于擬南芥根組織的單細胞轉錄組學研究報道[9]。來自德國圖賓根大學植物分子生物學中心的研究人員運用10X Genomics 單細胞轉錄組測序方法對擬南芥的根組織細胞進行了圖譜鑒定。該圖譜提供了詳細的時空信息,確定了所有的主要細胞類型,包括靜止中心(QC細胞)的稀有細胞,揭示了在細胞命運轉化為獨特的細胞形狀和功能過程中的關鍵發育調控因子和下游基因。通過擬時間序列分析,該研究描繪了從細胞從niche到分化的精細發生軌跡及主要調控轉錄因子。幾乎在同一時間,華盛頓大學基因組科學中心的研究人員同樣利用擬南芥根組織的單細胞測序研究在植物頂級期刊《Plant Cell》上發表了擬南芥根圖譜結果[10]。在該研究中,除了和上述兩篇文章類似的根部細胞分類之外,還通過熱脅迫處理,來揭示在非生物脅迫下細胞內部的響應異質性。該研究表面單細胞轉錄組學研究在植物發育和生理學研究中都有這廣闊的前景。
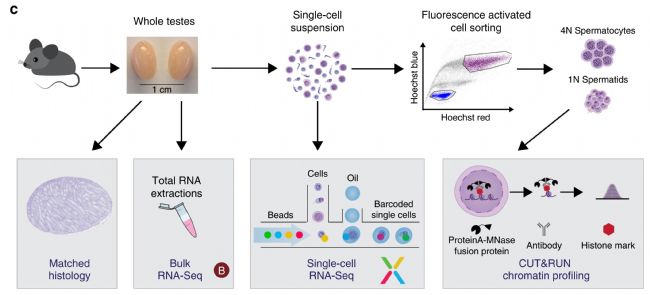
精子生成過程是一個復雜又具有嚴格分化過程的生理過程,繼18年10月份《Cell Research》上的成年人睪丸轉錄組圖譜研究[1]和19年2月份《Cell Reports》上新生與成年人睪丸細胞單細胞水平鑒定分類[2]的步伐,本月在《Nature Communications》上,歐洲生物信息學研究所歐洲分子生物學實驗室的研究人員用10x Genomics平臺對成年和幼年發育期間小鼠的睪丸進行了單細胞轉錄組測序和分析[3]。該研究精確分類了生殖細胞發育不同階段的細胞,包括稀有體細胞和精原細胞。值得一提的是,為精確捕獲低轉錄活性的細胞類型,研究人員應用一種新的統計工具EmptyDrops,識別到了先前研究沒有發現的細線期和偶線期精母細胞。此外,通過CUR和RUN實驗,研究人員對減數分裂后X染色體重新激活的時間動態模式進行了分析,確定了減數分裂后經歷了大量的染色質重塑現象。該研究為理解精子生成過程中的復雜過程及重要事件提供了新的認識。

圖1實驗設計線路
2.單細胞測序用于免疫研究多點開花
(1)Memory CD4+ T cells研究
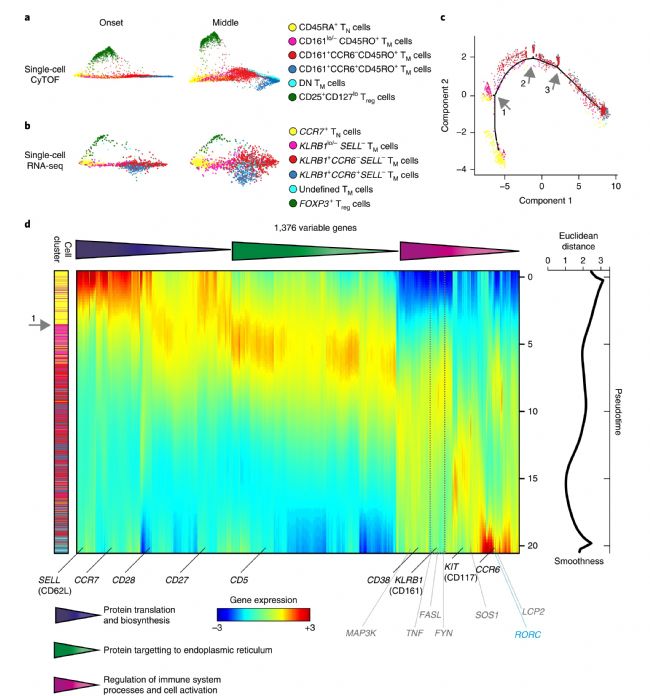
適應性免疫是建立在從克隆多樣的幼稚前體庫中選擇和擴展抗原特異性T細胞基礎上,但我們對于早期適應性免疫的認識還很有限。本月在《Nature Immunology》上,來自萊頓大學醫學中心免疫血液學與輸血學系的研究人員,通過流式細胞分選,10x Genomics單細胞轉錄組和免疫組庫分析,鑒定到了胎兒腸道22種CD4+T細胞類型[4]。結果顯示,Memory-like CD4+T細胞高表達Ki-67。通路分析揭示了與細胞活化和促炎效應功能相關的分化軌跡。TCR譜系分析表明克隆擴增,不同的譜系特征和Memory-like CD4 + T細胞亞群之間的相互連接。流式細胞成像表明Memory-like CD4 + T細胞與抗原呈遞細胞共定位。該研究提供了人胎兒腸道中產生Memory-like CD4 + T細胞的證據。

圖2 胎兒腸道CD4 + T細胞的分化軌跡分析
(2)exhausted CD8+ T 細胞亞群差異介導腫瘤控制和免疫檢查點阻斷響應
T細胞功能障礙是許多癌癥的標志,但是T細胞功能障礙的基礎和抑制性受體PD-1(抗PD-1)的抗體阻斷再生T細胞的機制尚不完全清楚。本月在《Nature Immunology》上,來自Dana-Farber癌癥研究所兒科腫瘤科的研究人員通過10X Genomics 單細胞轉錄組測序等實驗,發現抗PD-1療法作用于exhausted CD8+ T腫瘤浸潤淋巴細胞(TIL)的特定亞群[5]。與慢性病毒感染類似,功能失調的CD8 + TIL具有經典的表觀遺傳和轉錄耗竭特征。包括“祖細胞耗盡”TILs在內的exhausted CD8+ T 細胞亞群,能保留多功能性,持續長期存在并分化為“最終耗竭”TILs。患有較高百分比的祖源耗盡細胞的黑色素瘤患者對免疫治療的反應持續時間較長。因此,擴大祖源耗盡的CD8 + T細胞群可能是改善對檢查點阻斷響應的重要方法。
(3)干細胞記憶樣T細胞的產生過程追蹤
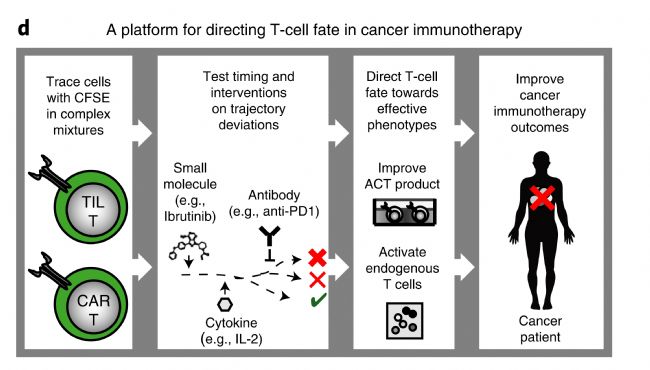
Naive T細胞選擇性分化成多能T細胞在臨床上對于基于細胞的癌癥免疫療法具有重要意義。在本月《Nature Biotechnology》上,來自斯坦福大學的研究人員,采用改良的染料稀釋法對Naive T細胞進行了增殖過程跟蹤[6]。利用23個marker,研究人員定義了主要由分裂狀態或者時間控制的一組蛋白,并發現未分裂的細胞占表型多樣性細胞的大部分。研究人員構建了Naive T細胞擴增過程中的細胞狀態變化圖譜。通過檢查該譜圖上的細胞信號,研究人員選擇了一種BTK和ITK抑制劑——ibrutinib,在T細胞分化之前給予處理以指導細胞分化成T單細胞記憶(TSCM)樣表型細胞。本研究的細胞命運追蹤方法為指導細胞分化提供了一種可行工具。

圖3 T細胞增殖跟蹤法用于癌癥免疫治療路線
3.單細胞轉錄組測序用于腦神經研究
下丘腦外側區域(LHA)協調一系列基本行為,包括睡眠,清醒,喂養,壓力和動機行為。在本月《Nature Neuroscience》上,來自康涅狄格大學生理學和神經生物學系的研究人員通過10X Genomics 單細胞轉錄組測序方法對小鼠LHA中分子不同細胞類型進行了分析和鑒定[7]。該研究共定義了15個不同谷氨酸能神經元群和15個GABAergic神經元,包括已知的和新的細胞類型。運用解剖學和行為學方法,研究人員進一步鑒定了一種生長抑素表達的新神經元群體,在先天運動行為中特異性表達。本研究為更好地理解LHA功能奠定了基礎。
下丘腦外側區域(LHA)協調一系列基本行為,包括睡眠,清醒,喂養,壓力和動機行為。在本月《Nature Neuroscience》上,來自康涅狄格大學生理學和神經生物學系的研究人員通過10X Genomics 單細胞轉錄組測序方法對小鼠LHA中分子不同細胞類型進行了分析和鑒定[7]。該研究共定義了15個不同谷氨酸能神經元群和15個GABAergic神經元,包括已知的和新的細胞類型。運用解剖學和行為學方法,研究人員進一步鑒定了一種生長抑素表達的新神經元群體,在先天運動行為中特異性表達。本研究為更好地理解LHA功能奠定了基礎。

圖4 LHAGlut和LHAGABA神經元細胞tSNE分類圖
4.單細胞轉錄組測序在植物領域嶄露頭角
相比動物的單細胞研究,由于植物具有細胞壁的特點,使得植物的單細胞研究相比較難,但是繼2月份的擬南芥根組織單細胞轉錄組學研究[8],本月在《Developmental Cell》上又發表了一篇基于擬南芥根組織的單細胞轉錄組學研究報道[9]。來自德國圖賓根大學植物分子生物學中心的研究人員運用10X Genomics 單細胞轉錄組測序方法對擬南芥的根組織細胞進行了圖譜鑒定。該圖譜提供了詳細的時空信息,確定了所有的主要細胞類型,包括靜止中心(QC細胞)的稀有細胞,揭示了在細胞命運轉化為獨特的細胞形狀和功能過程中的關鍵發育調控因子和下游基因。通過擬時間序列分析,該研究描繪了從細胞從niche到分化的精細發生軌跡及主要調控轉錄因子。幾乎在同一時間,華盛頓大學基因組科學中心的研究人員同樣利用擬南芥根組織的單細胞測序研究在植物頂級期刊《Plant Cell》上發表了擬南芥根圖譜結果[10]。在該研究中,除了和上述兩篇文章類似的根部細胞分類之外,還通過熱脅迫處理,來揭示在非生物脅迫下細胞內部的響應異質性。該研究表面單細胞轉錄組學研究在植物發育和生理學研究中都有這廣闊的前景。
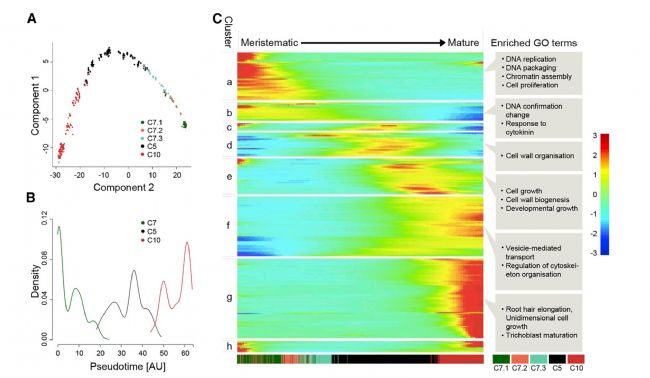
圖5 根細胞中分生到細胞成熟過程的基因變化熱圖
5.單細胞研究方法學進展
除了以上單細胞轉錄組學研究進展之外,基于單細胞組學分析的方法學研究在本月也有成果發表,其中包括《Nature Method》關于細胞成分分析的CPM方法[11],《Nucleic Acids Research》關于基于單細胞轉錄組數據的細胞特異性網絡構建[12],《Genome Biology》上關于基于液滴單細胞轉錄組數據中低轉錄水平細胞識別(EmptyDrops)軟件的開發[13],《Nature Biotechnology》上用于細胞可能性命運鑒定的Palantir算法開發[14]及《Nature Method》上有關tSNE算法優化的研究[15]。這些研究都為未來單細胞組學研究的進一步發展提供了強有力的工具。
為方便國內科研工作者的單細胞組學研究,上海生物芯片有限公司(SBC)于2018年引進10x Genomics單細胞組學檢測平臺,可為廣大科研工作者提供從樣本到分析的一體化解決方案,歡迎廣大科研朋友們與我們開展深入的溝通和廣泛的合作
- Guo, J., et al., The adult human testis transcriptional cell atlas. Cell Res, 2018. 28(12): p. 1141-1157.
- Sohni, A., et al., The Neonatal and Adult Human Testis Defined at the Single-Cell Level. Cell Rep, 2019. 26(6): p. 1501-1517 e4.
- Ernst, C., et al., Staged developmental mapping and X chromosome transcriptional dynamics during mouse spermatogenesis. Nat Commun, 2019. 10(1): p. 1251.
- Li, N., et al., Memory CD4(+) T cells are generated in the human fetal intestine. Nat Immunol, 2019. 20(3): p. 301-312.
- Miller, B.C., et al., Subsets of exhausted CD8(+) T cells differentially mediate tumor control and respond to checkpoint blockade. Nat Immunol, 2019. 20(3): p. 326-336.
- Good, Z., et al., Proliferation tracing with single-cell mass cytometry optimizes generation of stem cell memory-like T cells. Nat Biotechnol, 2019. 37(3): p. 259-266.
- Mickelsen, L.E., et al., Single-cell transcriptomic analysis of the lateral hypothalamic area reveals molecularly distinct populations of inhibitory and excitatory neurons. Nat Neurosci, 2019. 22(4): p. 642-656.
- Ryu, K.H., et al., Single-cell RNA sequencing resolves molecular relationships among individual plant cells. Plant Physiol, 2019.
- Denyer, T., et al., Spatiotemporal Developmental Trajectories in the Arabidopsis Root Revealed Using High-Throughput Single-Cell RNA Sequencing. Dev Cell, 2019. 48(6): p. 840-852 e5.
- Jean-Baptiste, K., et al., Dynamics of gene expression in single root cells of A. thaliana. Plant Cell, 2019.
- Frishberg, A., et al., Cell composition analysis of bulk genomics using single-cell data. Nat Methods, 2019.
- Dai, H., et al., Cell-specific network constructed by single-cell RNA sequencing data. Nucleic Acids Res, 2019.
- Lun, A.T.L., et al., EmptyDrops: distinguishing cells from empty droplets in droplet-based single-cell RNA sequencing data. Genome Biol, 2019. 20(1): p. 63.
- Setty, M., et al., Characterization of cell fate probabilities in single-cell data with Palantir. Nat Biotechnol, 2019.
- Linderman, G.C., et al., Fast interpolation-based t-SNE for improved visualization of single-cell RNA-seq data. Nat Methods, 2019. 16(3): p. 243-245.
標簽:
轉錄組單細胞領域研究
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com