微生物宏基因組學及后期數據分析專題培訓班通知
“微生物宏基因組學及后期數據分析專題培訓班”的通知
各有關單位:
微生物作為地球環境中的一大類生物群體,每種微生物都有其獨特的功能。微生物學(microbiology)作為生物學的分支學科之一,在許多學科領域都發揮著舉足輕重的作用。作為國內最大的綜合性微生物學研究機構,為推動我國微生物學的發展,提高從業人員的技術水平,更好地與微生物研究領域的專家學者們分享、交流在微生物研究領域中的心得、經驗。應廣大行業工作者的要求,由中科成創(北京)生物技術有限公司舉辦“生物信息學最新技術-微生物宏基因組學及后期數據分析專題培訓班。培訓班采用理論和演示相結合的教學形式,使學員在短時間內對微生物研究中涉及的知識和方法得到進一步地提高,同時學員與授課專家進行現場交流與咨詢,探討學員在平時工作、研究中的瓶頸問題。與同行交流以拓寬自己的研究思路,共同探討和挖掘微生物資源的科學研究價值和應用前景。具體事宜通知如下:
一、培訓目標及特點:
本培訓以微生物宏基因組學技術的應用與數據分析為主題,精心設計了具有前沿性、實用性和針對性強的理論課程和上機課程。培訓邀請的主講人均是有理論和實際研究經驗的人員。學員通過與專家直接交流,能夠分享到這些頂尖學術機構的研究經驗和實驗設計思路。學員通過集中專題學習后能夠擴展思路,在研究技術方面領悟更多。
二、授課專家:
主講專家來自中科院科研機構的高級專家,擁有豐富的科研工作經驗,長期從事生物信息學方面的項目研究,發表Nature, Nature Cell Biology, Molecular Cell等雜志40多篇。具有資深的技術底蘊和專業背景,目前承擔國家科技部、國家自然基金委和市科技新星項目等多項課題。
三、培訓對象:
大中專院校生物信息、生物計算、生命科學、醫學、化學、農學、計算機科學、數學類專業的課程負責人、一線教師、教研室骨干人員、教學管理人員;科研單位從事生物、生命科學、微生物研究的相關人員;生物、醫藥、化學及相關企業的領導與技術骨干。
四、時間地點: 2020年12月10日—12月13日 江蘇 南京 (時間安排:第1天報到,授課3天)
五、培訓內容:見附件二
六、報名辦法及費用:
每人¥3980元(含報名費、培訓費、資料費、上機費)
團隊報名:4+1優惠。(5位其中1位免除費用)
食宿可統一安排,費用自理。各單位人員報名參加,報名回執表請回傳至會務處即可。
如單位有內訓需求,請將內訓方案傳至會務組,根據單位需求安排相應專家進行授課
七、聯系方式:
聯系人: 張琦 17744569660
報名郵箱:zkcc_BIS@vip.163.com
中科成創(北京)生物技術有限公司
二零二零年十月二十六日
附件一:報名回執表二零二零年十月二十六日
附件二:培訓內容
| (12月11日 上午) 微生物基因組 |
微生物基因組和轉錄組學研究 1.1、微生物基因組研究的意義 1.2、微生物基因組研究概況 1.3、微生物基因組的特點 1.4、微生物轉錄組學研究 |
| (12月11日 下午) R語言繪圖 |
R語言基本介紹 R語言基本運算、向量函數 1.3 R語言數據讀入以及導出 ggplot2基本繪圖 (條形圖、散點圖、折線圖等) |
| (12月12日 上午) 宏基因組學 |
1. 16S rDNA AMPLICON SEQUENCING 法 1.1. 測序平臺、引物設計及區域選擇不同測序平臺、針對細菌、真菌不同微生物實驗設計 1.2. 測序量及采樣建議 針對水體、糞便、土壤、物體表面、口腔等不同生境 1.3. 分析流程圖 數據收集、數據預處理、數據分析 1.4. 結果解析 1.4.1 OTU聚類 1.4.2 物種注釋 1.4.3 物種分布情況 1.4.4 樣品復雜度分析:α多樣性 Coverage Chao指數 ACE指數 Shannon曲線 Richness rarefaction曲線 1.4.5 多樣品比較分析:β多樣性 樣品間物種豐度熱圖 排序分析: PCA分析 PCoA分析 NMDS分析 Unifrac分析 樣品聚類分析 2. DSS (direct shotgun sequencing)法 2.1. 分析流程 2.2. 功能注釋分析 2.3. 代謝途徑解析 |
| (12月12日 下午) 微生物基因組及宏轉錄組常用軟件介紹 |
2.1 微生物基因組在線圈圖分析 2.2 代謝通路分析(KEGG & KAAS) 2.3 病原菌耐藥基因鑒定(CARD & Resfinder) 2.4 細菌基因組島鑒定 (Islandviewer) 2.5 CAZy 碳水化合物活性酶注釋(宏轉錄組) 2.6 基因差異表達分析(宏轉錄組) 2.7 基因聚類分析(宏轉錄組) |
| (12月13日 上午) 宏轉錄組學 |
1 宏轉錄組學介紹 2 宏轉錄組學定義 3 宏轉錄組研究方法 4 宏轉錄組研究內容 5 宏基因組與宏轉錄組的差別 |
| (12月13日 下午) 16S測序數據分析及宏基因組學軟件介紹 |
1. 筆記本電腦配置要求建議筆記本內存4G以上,64位操作系統 2. 針對16S rDNA amplicon sequencing分析 2.1. 虛擬機安裝:Virtual Box 2.2. 16S分析運行環境搭建:QIIME Virtual Machine 2.3. 實例演示及結果展示 數據預處理 OTU 聚類 物種注釋 OTU table生成 α多樣性分析 序列比對 構建進化樹 β多樣性分析 3. 針對shotgun sequencing分析(只做流程演示講解) 3.1. 序列質量控制:fastqc 3.2. 序列拼接 3.3. 基因預測及豐度分析 3.4. 物種注釋 3.4. 功能注釋 3.5. MetaSPAdes、MEGAN、metAMOS等軟件介紹 |
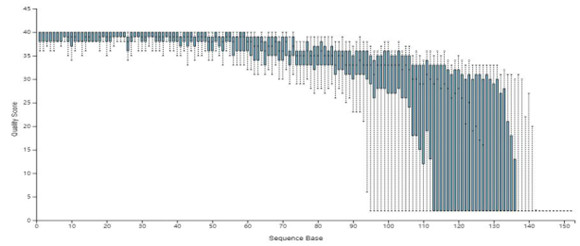
Figure 1 QIIME2結果1:16s rDNA測序數據質控信息
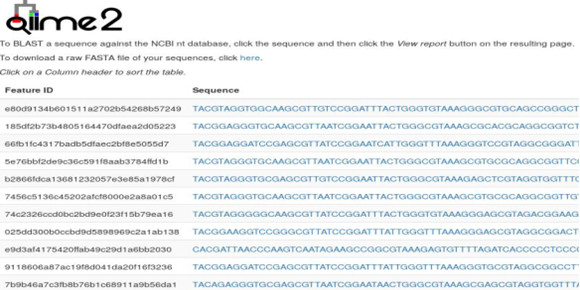
Figure 2 QIIME2結果2:features的序列信息
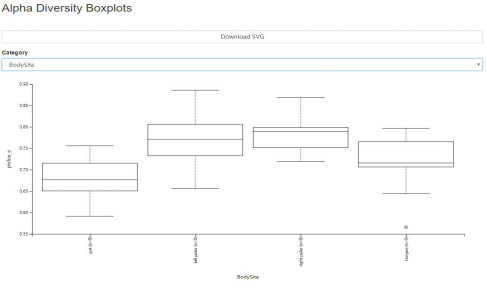
Figure 3 QIIME2結果3:alpha diversity 差異分析
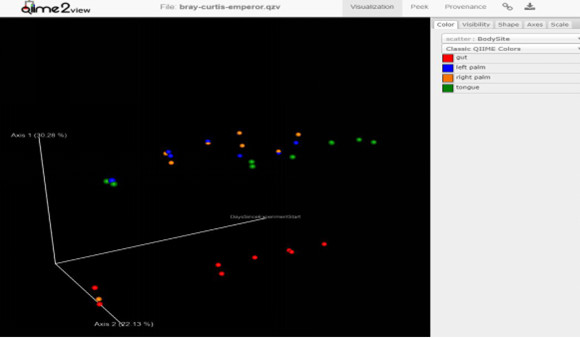
Figure 4 QIIME2結果3:beta diversity 可視化
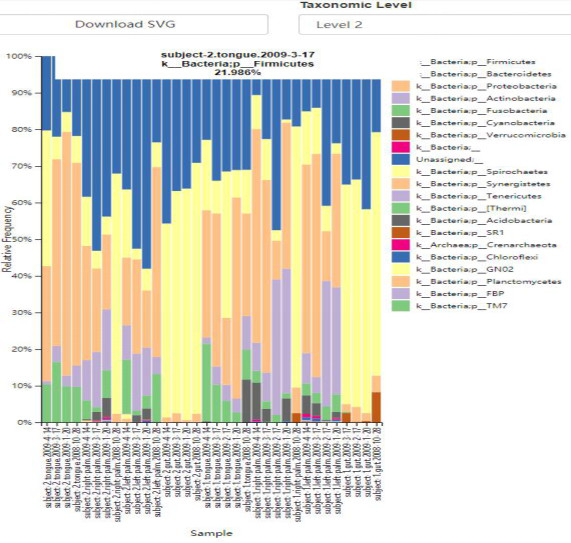
Figure 5 QIIME2結果5:物種注釋結果可視化
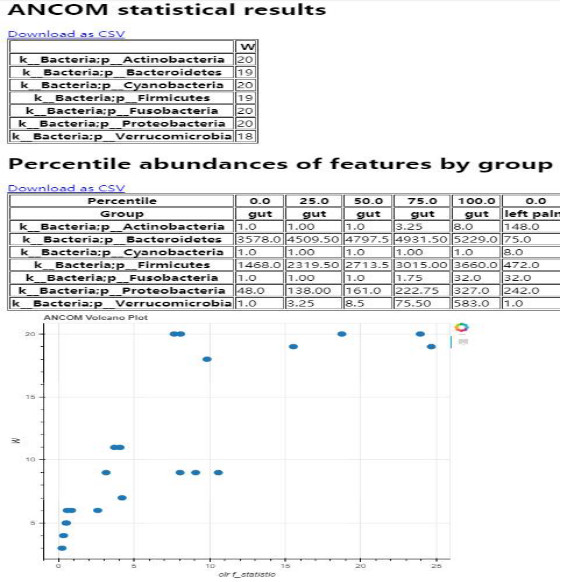
Figure 6 QIIME2結果6:差異菌群分析結果
- 講座邀請:Savante SEND標準數據集自動化解決方案
- 東樂自然基因第72屆膜片鉗線下實際操作培訓班通知
- 易科泰受邀參加全國土壤環境生物安全與健康研討會
- 斯高&埃德講座預告:心臟科研中常用電生理技術及進展
- 2025第四期新酶設計及酶技術應用專題培訓班通知
- 易科泰受邀參加第三屆“千種本草基因組計劃”研討會
- 進科馳安第四場BMG多功能酶標儀線上答疑會邀請函
- 2025生物人PCR產業技術深度研討學習班報名通知
- 講座邀請:AI力場到量子力學,FEP工作流自動化
- 易科泰邀您參加全國土壤環境生物安全與健康研討會
- 萊闊邀您參加Harvesting Carbon 作者見面會
- 講座:高通量多參數分析系統在線粒體研究中的應用
- 易科泰邀您參加第三屆“千種本草基因組計劃”研討會
- 怡美通德誠邀您參加CCTB 2025腫瘤標志物學術大會
- 直播:共聚焦熒光壽命成像在植物相關研究中的應用
- 第五屆多組學科研與臨床應用大會五一福利大放送
- ATMP 2025第九屆先進療法創新峰會通知
- 安捷倫在AACR 2025展示開創性解決方案與合作成果
- 10x Genomics技術方案更新(3月),新手冊邀您下載
- 安捷倫胃腺癌等伴隨診斷檢測產品獲得歐盟IVDR認證
- 森西賽智亮相中國細胞生物學學會2025年全國大會
- 森西賽智科技受邀亮相第七屆病毒腫瘤學學術會議
- 怡美通德誠邀您參加CCTB 2025腫瘤標志物學術大會
- 中山大學王鵬教授榮獲安捷倫ACT-UR研究基金支持
- 森西賽智聯合Vizgen在兩場大會正式推出MERFISH2.0
- 怡美通德邀您參加第三屆國際單細胞及空間組學大會
- 10x Genomics技術方案更新(2月),新手冊等您獲取
- 2025中國細胞與基因治療大會第五屆年會招展通知
- 10x和CZI及Ultima Genomics合作啟動十億細胞項目
- 第二屆中國國際生殖醫學健康大會通知
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com






